使用 Dask Array 在集群上分布式 NumPy
这项工作得到 Continuum Analytics、XDATA 项目以及 摩尔基金会数据驱动发现计划的支持
本页包含嵌入的大型配置文件。它在实际网站上可能看起来更好,而不是通过像 planet.python 这样的联合页面,并且在非宽带连接上加载可能需要一段时间(总大小约为 20MB)
总结
我们使用 Dask array 在 Amazon EC2 上的机器集群中并行分析分布式的 NumPy 数组堆栈图像。这是一个在许多图像分析组(从卫星图像到生物医学应用)之间共享的典型应用。我们将经历一系列常见操作
- 使用 Scikit Image 在本地检查图像样本
- 围绕所有图像构建一个分布式的 Dask.array
- 使用 Numba 处理图像并重新居中
- 转置数据以获得每个像素的时间序列,计算 FFTs
最后一步非常有趣。即使您只是浏览本文的其余部分,我也建议您查看最后一部分。
检查数据集
我向美国国立卫生研究院 (NIH) 的一位同事询问了一个相当大的图像数据集。他回复了以下信息
电子显微镜可能是该领域生成最大 ndarray 数据集的仪器——通常是数 TB 级别。神经科学需要电镜来观察神经元之间的连接,因为神经突触(连接)的关键特征低于光学显微镜的衍射极限。这种类型的研究被称为“连接组学”。许多团队正在研究机器视觉方法,以跟踪从小神经元部分从一个切片到下一个切片。
这些数据来自果蝇:http://emdata.janelia.org/。这里是数据的一个 2D 切片示例 http://emdata.janelia.org/api/node/bf1/grayscale/raw/xy/2000_2000/1800_2300_5000。
import skimage.io
import matplotlib.pyplot as plt
sample = skimage.io.imread('http://emdata.janelia.org/api/node/bf1/grayscale/raw/xy/2000_2000/1800_2300_5000'
skimage.io.imshow(sample)
URL 中的最后一个数字是大约 10000 张图像堆栈中的索引。我们可以更改该数字以获取我们 3D 数据集中的不同切片。
samples = [skimage.io.imread('http://emdata.janelia.org/api/node/bf1/grayscale/raw/xy/2000_2000/1800_2300_%d' % i)
for i in [1000, 2000, 3000, 4000, 5000, 6000, 7000, 8000, 9000]]
fig, axarr = plt.subplots(1, 9, sharex=True, sharey=True, figsize=(24, 2.5))
for i, sample in enumerate(samples):
axarr[i].imshow(sample, cmap='gray')
我们看到感兴趣的区域随时间在帧中移动,并在开始和结束时减弱。
创建分布式数组
尽管我们的数据分散在许多文件中,但我们仍然希望将其视为一个单一的逻辑 3D 数组。我们知道如何使用 Scikit-image 获取该数组的任何特定 2D 切片。现在我们将使用 Dask.array 将所有这些 Scikit-image 调用拼接成一个单一的分布式数组。
import dask.array as da
from dask import delayed
imread = delayed(skimage.io.imread, pure=True) # Lazy version of imread
urls = ['http://emdata.janelia.org/api/node/bf1/grayscale/raw/xy/2000_2000/1800_2300_%d' % i
for i in range(10000)] # A list of our URLs
lazy_values = [imread(url) for url in urls] # Lazily evaluate imread on each url
arrays = [da.from_delayed(lazy_value, # Construct a small Dask array
dtype=sample.dtype, # for every lazy value
shape=sample.shape)
for lazy_value in lazy_values]
stack = da.stack(arrays, axis=0) # Stack all small Dask arrays into one
>>> stack
dask.array<shape=(10000, 2000, 2000), dtype=uint8, chunksize=(1, 2000, 2000)>
>>> stack = stack.rechunk((20, 2000, 2000)) # combine chunks to reduce overhead
>>> stack
dask.array<shape=(10000, 2000, 2000), dtype=uint8, chunksize=(20, 2000, 2000)>
因此,在这里我们通过对 skimage.io.imread 的 10000 个延迟调用构建了一个惰性 Dask.array。我们还没有做任何实际工作,我们只是构建了一个并行数组,它知道如何通过在必要时下载正确的图像来获取任何特定数据切片。这为所有这些远程图像提供了一个完整的 NumPy 式抽象。例如,现在只需对 Dask 数组进行切片即可下载特定的图像。
>>> stack[5000, :, :].compute()
array([[0, 0, 0, ..., 0, 0, 0],
[0, 0, 0, ..., 0, 0, 0],
[0, 0, 0, ..., 0, 0, 0],
...,
[0, 0, 0, ..., 0, 0, 0],
[0, 0, 0, ..., 0, 0, 0],
[0, 0, 0, ..., 0, 0, 0]], dtype=uint8)
>>> stack[5000, :, :].mean().compute()
11.49902425
然而,在连接到集群之前,我们可能不希望进行太多进一步操作。这样我们就可以一次性将所有图像下载到分布式内存中,并开始进行一些实际计算。我碰巧在 Amazon EC2 上有十台 m4.2xlarge 实例(每台 8 核,30GB RAM)运行 Dask worker。所以我们将连接到这些实例。
from dask.distributed import Client, progress
client = Client('schdeduler-address:8786')
>>> client
<Client: scheduler="scheduler-address:8786" processes=10 cores=80>
我用 `scheduler-address` 替换了我的调度器的实际地址(例如 54.183.180.153)。现在让我们把所有的图像加载进来,将数组持久化到内存中的具体数据。
stack = client.persist(stack)
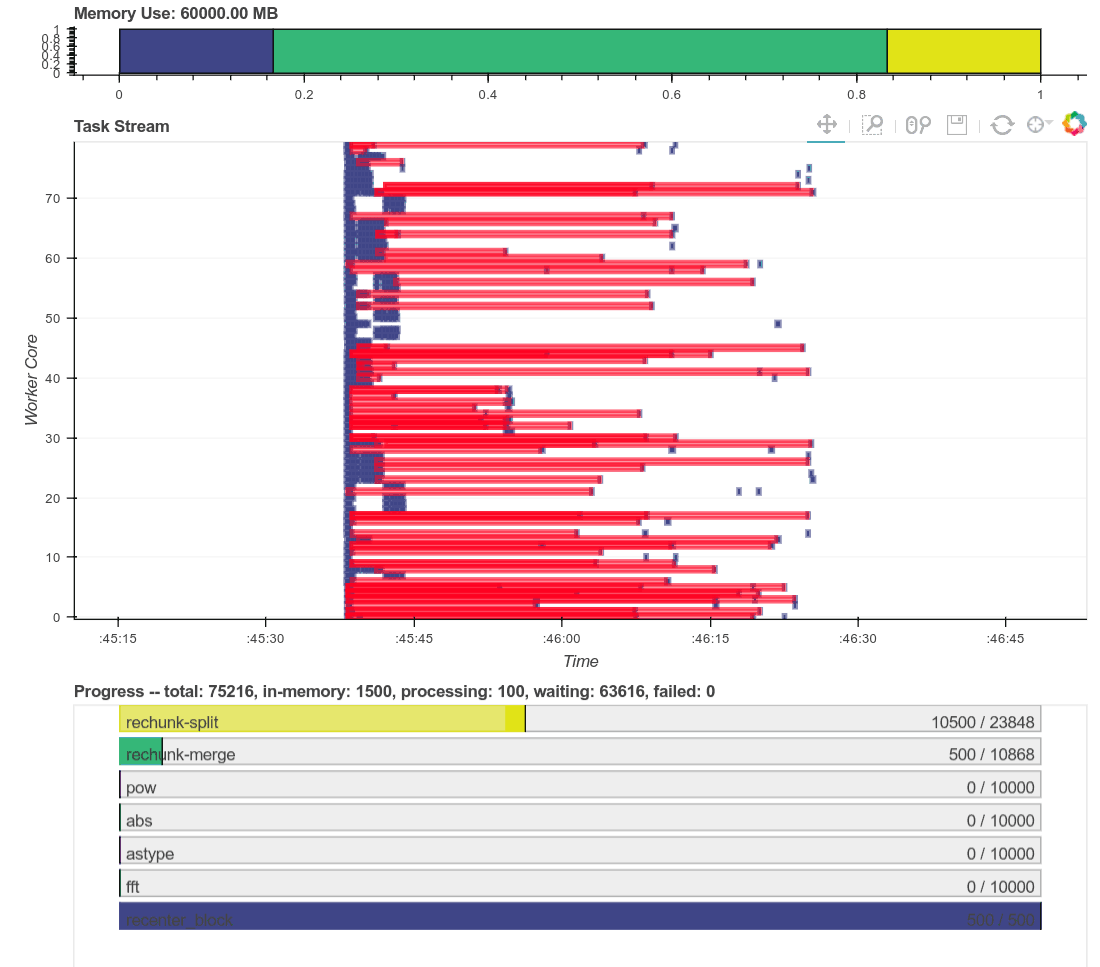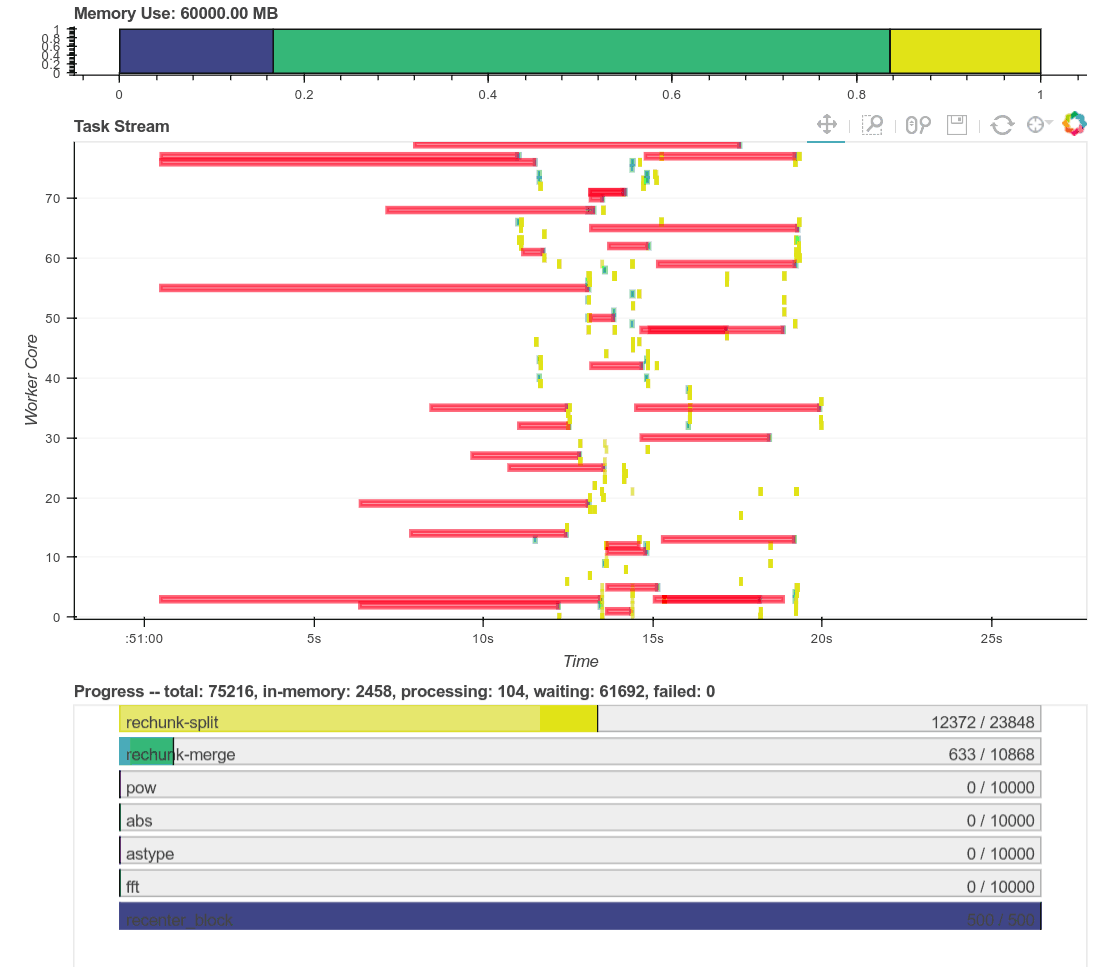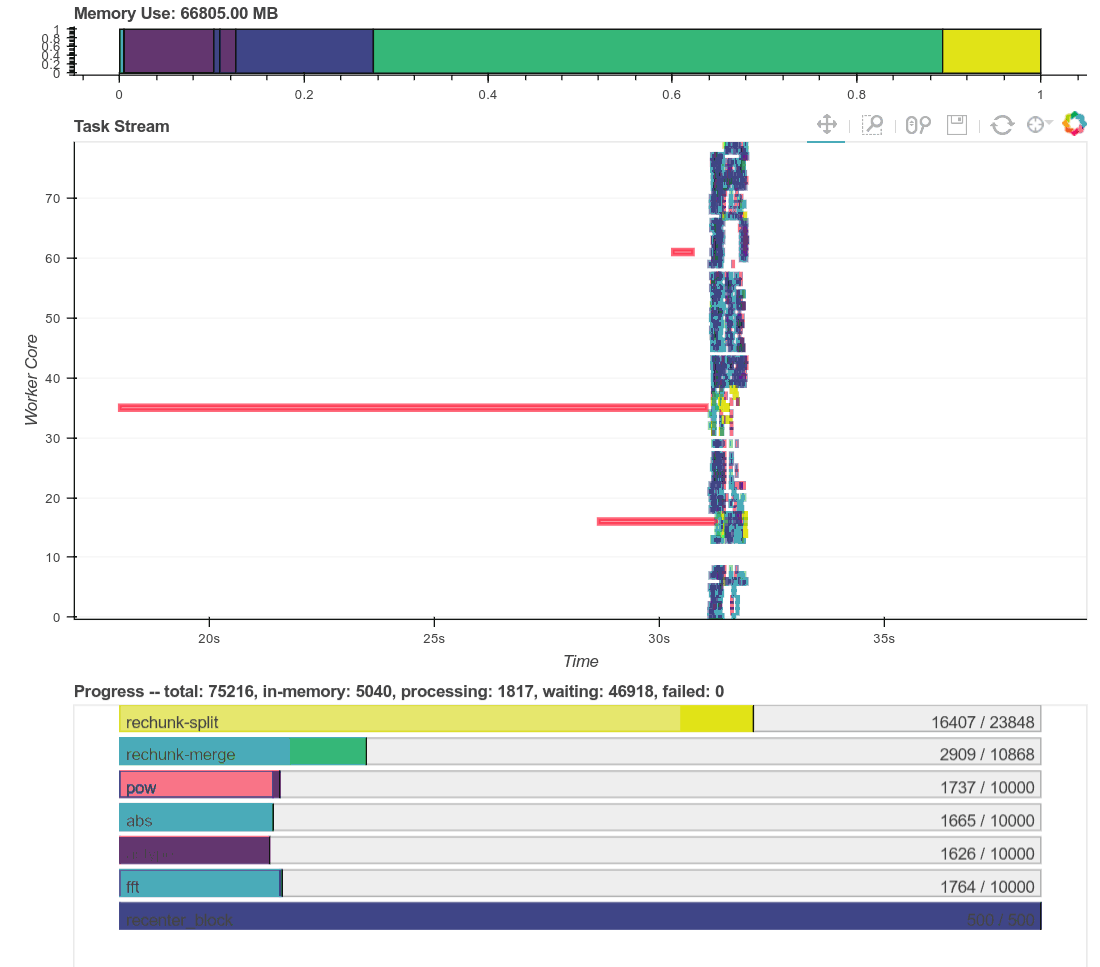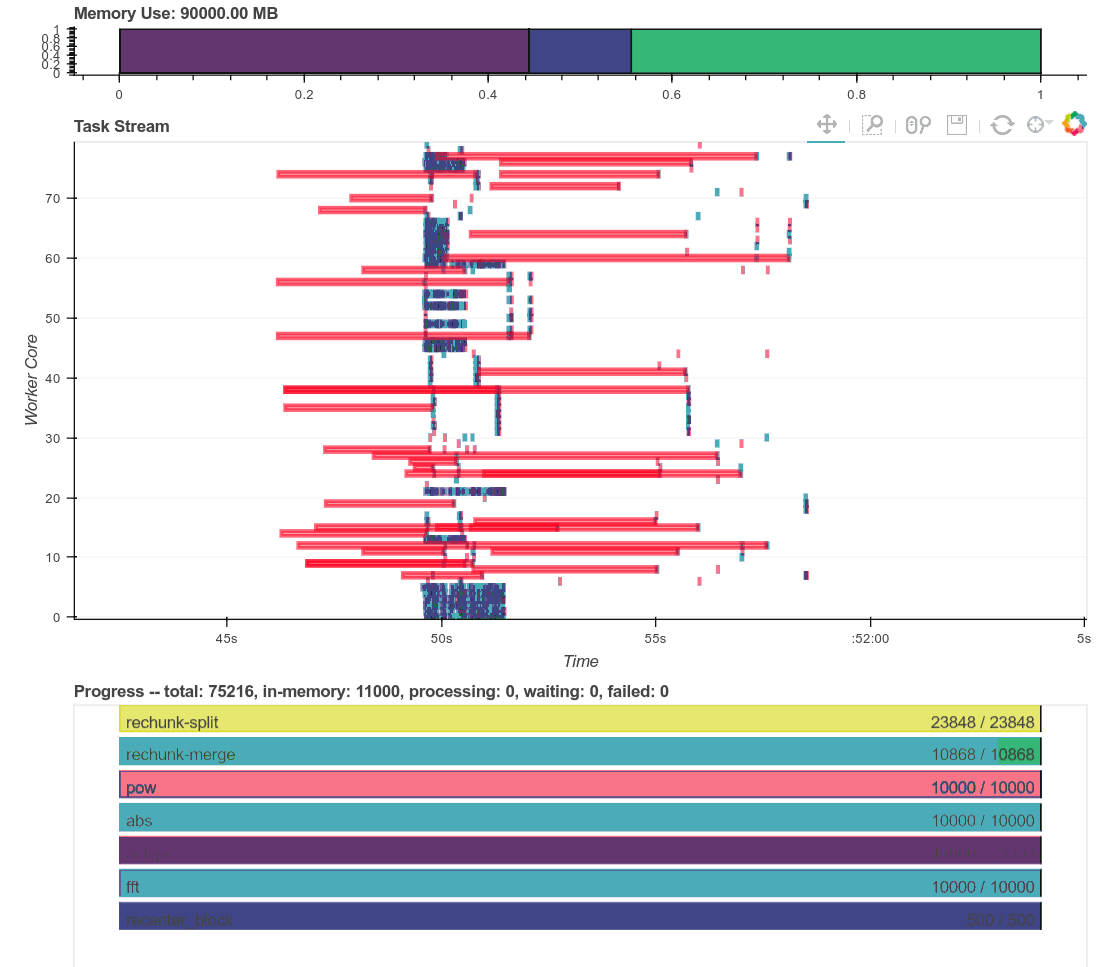
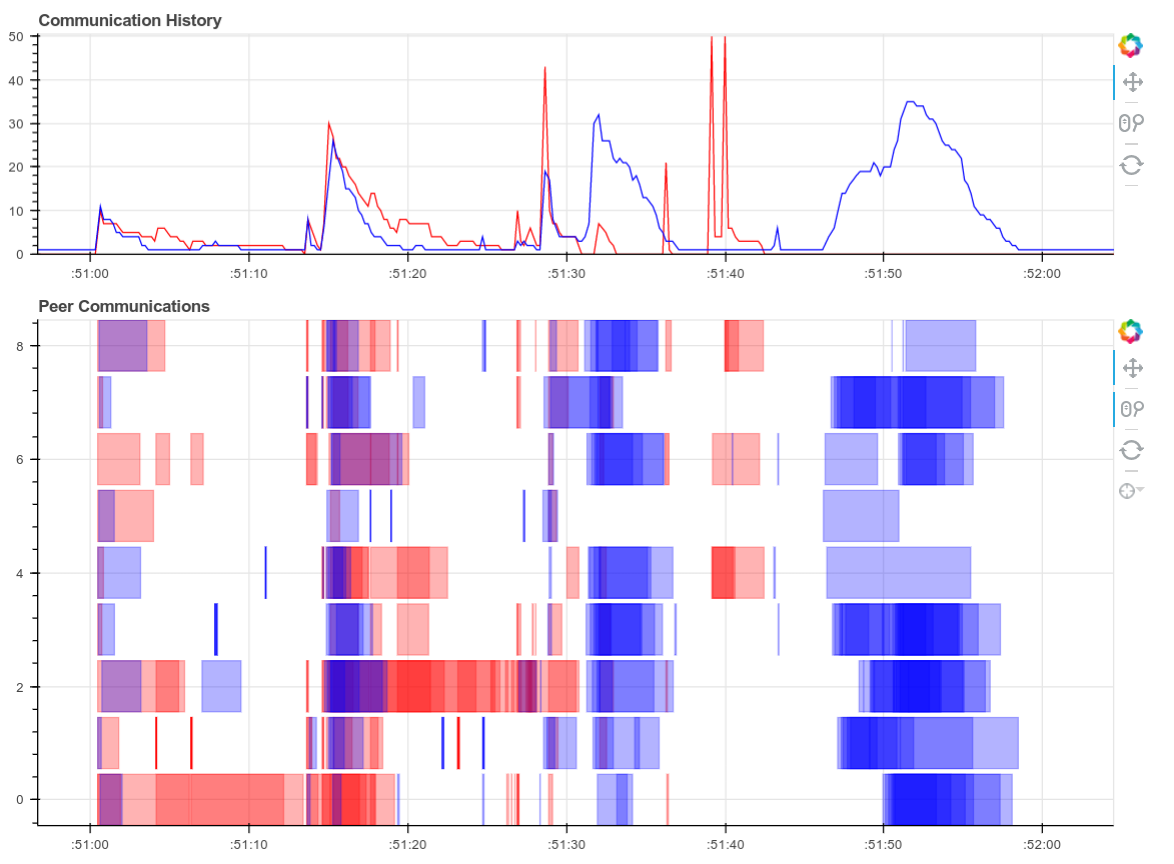
这开始在我们的 10 个 worker 上下载 10000 张图像。完成后,我们有 10000 个 NumPy 数组分散在我们的集群中,由我们单一的逻辑 Dask 数组协调。这需要一些时间,大约五分钟。这里主要受网络带宽限制(Janelia 的服务器与我们的计算节点不在同一位置)。这里是计算的并行配置文件,它是一个交互式的 Bokeh 图。
在这篇博文中将有几个这样的配置文件图,所以您现在可能想熟悉它们。此图中的每个水平矩形都对应于我们集群中某个地方随时间运行的单个 Python 函数。由于我们调用了 skimage.io.imread 10000 次,因此有 10000 个紫色矩形。它们沿 y 轴的位置表示它们在我们集群的 80 个核心中的哪个核心上运行,而它们沿 x 轴的位置表示它们的开始和停止时间。您可以将鼠标悬停在每个矩形(函数)上,以获取有关任务类型、花费时间等更多信息。在下图中,紫色矩形是 skimage.io.imread 调用,红色矩形是集群中 worker 之间的数据传输。单击图像右上角的放大镜图标以启用缩放工具。
现在我们将 Dask 数组持久化到内存中,我们的数据基于集群中数百个具体的内存中的 NumPy 数组,而不是基于数百个惰性的 scikit-image 调用。现在我们可以更快地进行各种有趣的分布式数组计算。
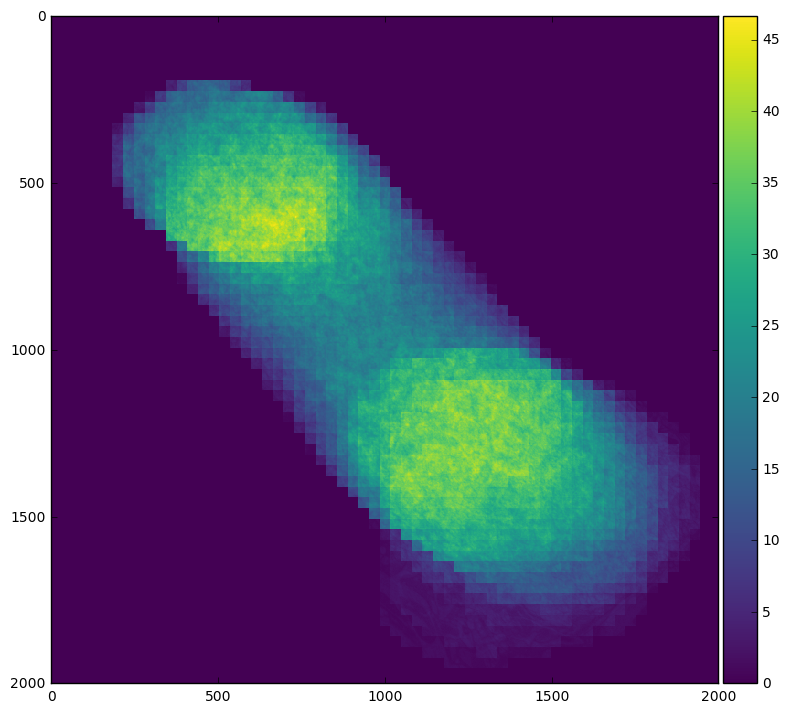
例如,我们可以通过对时间轴求平均来轻松看到感兴趣的区域在帧中移动
skimage.io.imshow(stack.mean(axis=0).compute())
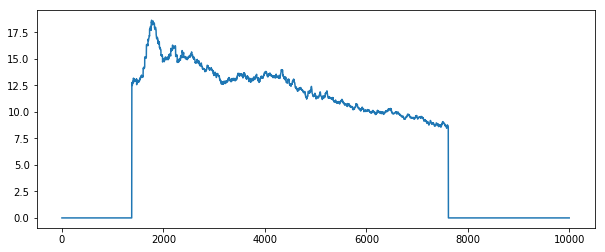
或者我们可以通过对 x 和 y 轴求平均来查看感兴趣的区域实际何时出现在帧内
plt.plot(stack.mean(axis=[1, 2]).compute())
通过查看每种情况下的配置文件图,我们可以看到对时间轴求平均涉及更多的节点间通信,在这种情况下这可能非常昂贵。
使用 Numba 重新居中图像
为了消除随时间变化的空间偏移,我们将计算每个切片的质心,然后围绕该中心裁剪图像。我在 Scikit-Image 文档中查找了质心,发现了一个功能远超我所需的函数,所以我只是快速地用纯 Python 编写了一个解决方案,然后使用 Numba 进行了 JIT 编译(这使得运行速度接近 C 语言)。
from numba import jit
@jit(nogil=True)
def centroid(im):
n, m = im.shape
total_x = 0
total_y = 0
total = 0
for i in range(n):
for j in range(m):
total += im[i, j]
total_x += i * im[i, j]
total_y += j * im[i, j]
if total > 0:
total_x /= total
total_y /= total
return total_x, total_y
>>> centroid(sample) # this takes around 9ms
(748.7325324581344, 802.4893005160851)
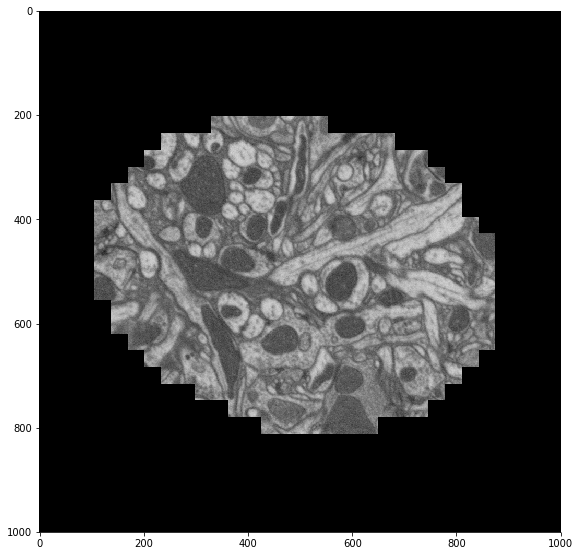
def recenter(im):
x, y = centroid(im.squeeze())
x, y = int(x), int(y)
if x < 500:
x = 500
if y < 500:
y = 500
if x > 1500:
x = 1500
if y > 1500:
y = 1500
return im[..., x-500:x+500, y-500:y+500]
plt.figure(figsize=(8, 8))
skimage.io.imshow(recenter(sample))
现在我们将此函数映射到我们的分布式数组上。
import numpy as np
def recenter_block(block):
""" Recenter a short stack of images """
return np.stack([recenter(block[i]) for i in range(block.shape[0])])
recentered = stack.map_blocks(recenter,
chunks=(20, 1000, 1000), # chunk size changes
dtype=a.dtype)
recentered = client.persist(recentered)
这个配置文件提供了一个很好的机会来谈论调度器失败;这里出了点小问题。一开始我们快速地重新居中了几张图像(Numba 速度很快),每组二十张图像大约需要 300-400ms。然而,当一些 worker 完成所有分配的任务后,调度器错误地开始负载均衡,将图像从繁忙的 worker 移动到空闲的 worker。不幸的是,此时的网络似乎比预期慢得多,因此移动 + 在别处计算的策略最终比让繁忙的 worker 完成工作慢得多。调度器精确地跟踪预期的计算时间和传输时间,以避免像这样错误。这类问题很少见,但偶尔也会发生。
我们通过对重新居中的图像沿时间轴求平均并将其显示到屏幕上来检查我们的工作。我们看到图像彼此之间更好地居中,正如预期的那样。
skimage.io.imshow(recentered.mean(axis=0))
这展示了使用 Numba 创建快速内存代码然后使用 Dask.array 进行扩展是多么容易。这两个项目很好地互补,使我们能够在集群上以直观的代码获得接近最优的性能。
按像素重新分块为时间序列
我们现在将把数据从按时间切片划分重新排列为按像素划分。这将使我们能够有效地对每个时间序列运行快速傅里叶变换 (FFT) 等计算。像这样来回切换分块模式对于分布式数组来说通常是非常困难的操作,因为数组的每个切片都贡献给每个时间序列。我们存在 N 平方通信。
此分析可能不适用于这些数据(我们不会从中获得任何有用的科学知识),但它代表了一个非常常见的问题,所以我想将其包含在内。
目前我们的 Dask 数组的分块形状是 (20, 1000, 1000),这意味着我们的数据被收集到集群中的 500 个 NumPy 数组中,每个数组的大小为 (20, 1000, 1000)。
>>> recentered
dask.array<shape=(10000, 1000, 1000), dtype=uint8, chunksize=(20, 1000, 1000)>
但我们想改变这种形状,使分块覆盖整个第一个轴。我们希望任何特定像素的所有数据都位于同一个 NumPy 数组中,而不是分散在数百个不同的 NumPy 数组中。我们可以通过重新分块来解决这个问题,使每个像素成为自己的块,如下所示
>>> rechunked = recentered.rechunk((10000, 1, 1))
然而,这会导致一百万个分块(有一百万个像素),这将导致一些调度开销。相反,我们将把我们的时间序列收集到由一百个像素组成的 10 x 10 组中。这将帮助我们减少开销。
>>> # rechunked = recentered.rechunk((10000, 1, 1)) # Too many chunks
>>> rechunked = recentered.rechunk((10000, 10, 10)) # Use larger chunks
现在我们计算每个像素的 FFT,取绝对值并平方以获得功率谱。最后,为了节省空间,我们将 dtype 降级到 float32(我们的原始数据无论如何也只有 8 位)。
x = da.fft.fft(rechunked, axis=0)
power = abs(x ** 2).astype('float32')
power = client.persist(power, optimize_graph=False)
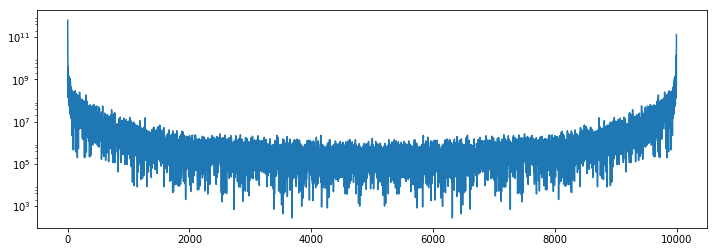
这是一个有趣的配置文件,值得检查;它包含了重新分块和随后的 FFTs。我们包含了执行期间的实时轨迹、完整的配置文件以及单个 worker 的一些诊断图。这些图的总大小约为 20MB。对于没有宽带访问的人们,我深表歉意。
这里是计算随时间完成的实时图
这里是计算完成后整个计算的单个交互式图。使用右上角的工具进行缩放。将鼠标悬停在矩形上以获取更多信息。记住,红色表示通信。
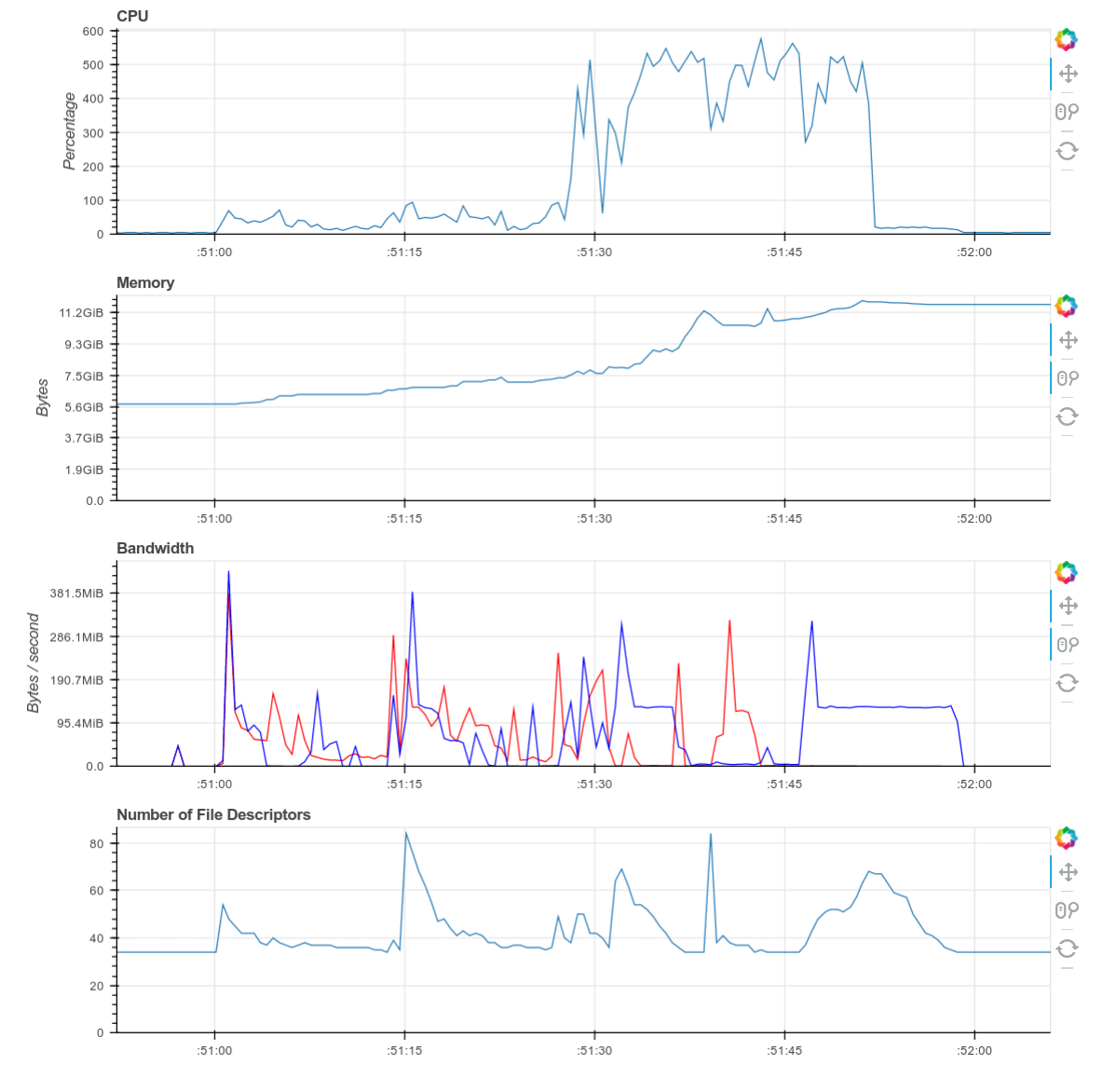
本次计算期间单个 worker 诊断仪表板的截图。
这个计算始于大量的通信,在我们重新分块和重新对齐数据时(Antoine Pitrou 在 dask #417 中的最新优化)。然后我们过渡到执行数千个小的 FFT 和其他算术操作。上面所有的图都显示了一个很好的过渡,从大量的通信到大量的处理,并且在两个方向都有一些重叠(一旦一些复杂的分块可用,我们就可以开始重叠通信和计算)。worker 之间的通信速度约为 100-300 MB/s(这是 Amazon EC2 的典型速度),并且 CPU 负载保持很高。我们正在充分利用我们的硬件。
最后我们可以检查结果。我们看到图像角落的功率谱非常单调,而在图像中心附近则有典型的活动。
plt.semilogy(1 + power[:, 0, 0].compute())
plt.semilogy(1 + power[:, 500, 500].compute())
最后思考
这篇博文展示了一个非平凡的图像处理工作流程,强调了以下几点
- 从惰性 SKImage 调用构建 Dask 数组。
- 结合 Dask.array 使用 NumPy 语法在集群上聚合分布式数据。
- 使用 Numba 构建质心函数。结合使用 Numba 和 Dask 来清理图像堆栈。
- 重新分块以促进时间序列操作。执行 FFTs。
希望这个例子包含的组件看起来与您希望在您的硬件上使用数据执行的操作相似。我们乐于看到更多像这样的实际应用。
我们可以做得更好的地方
像所有专注于计算的博文一样,我们也会包含一个关于出了什么问题以及如果有更多时间我们可以做得更好的部分。
- 通信成本过高: worker 间的通信本应只需要 200 毫秒,但却耗时 10 到 20 秒。我们需要更仔细地检查我们的通信管道(在其他计算中通常表现良好),看看是否有什么异常。讨论在此 dask/distributed #776,初步工作在此 dask/distributed #810。
- 负载均衡故障:我们发现了一个案例,我们的负载均衡启发式算法表现异常,错误地在 worker 之间移动数据,而实际上最好是让一切保持原样。这很可能是由于上面观察到的异常低的带宽问题造成的。
- 从磁盘加载阻塞网络 I/O:在进行此操作时,我们发现了一个问题,即从磁盘加载大量数据可能会阻塞 worker 对网络请求的响应(dask/distributed #774)
- 更大的数据集:在一个更大的数据集上尝试这个方案会很有趣,看看这里的解决方案如何扩展。
博客评论由 Disqus 提供