Dask 和 __array_function__ 协议 NEP-18 的进展
作者:Peter Andreas Entschev
摘要
Dask 在分析并行性方面用途广泛,但要将其应用于更广泛的领域仍然存在一个问题:使其能够透明地与类似 NumPy 的库一起工作。我们之前讨论过如何使用 GPU Dask 数组,但这仅限于共享类似 NumPy 接口的数组成员方法的范围,例如 .sum() 方法,因此,仍然无法调用 NumPy 库中的通用功能。NumPy 最近在 NEP-18 中通过引入 __array_function__ 协议解决了这个问题。简而言之,该协议允许 NumPy 函数调用根据输入的数组类型分派适当的类似 NumPy 库实现,从而允许 Dask 对此类库保持无知,内部仅调用 NumPy 函数,该函数会自动处理适当库实现的分派,例如 CuPy 或 Sparse。
为了理解这一改变的最终目标是什么,请考虑以下示例
import numpy as np
import dask.array as da
x = np.random.random((5000, 1000))
d = da.from_array(x, chunks=(1000, 1000))
u, s, v = np.linalg.svd(d)
现在考虑我们想要加速 Dask 数组的 SVD 计算并将该工作卸载到支持 CUDA 的 GPU 上,我们最终想要简单地用 CuPy 数组替换 NumPy 数组 x,并让 NumPy 通过 __array_function__ 协议发挥其魔力,并在底层分派适当的 CuPy 线性代数操作
import numpy as np
import cupy
import dask.array as da
x = cupy.random.random((5000, 1000))
d = da.from_array(x, chunks=(1000, 1000))
u, s, v = np.linalg.svd(d)
我们可以对 Sparse 数组或任何其他支持 __array_function__ 协议以及我们尝试执行的计算的类似 NumPy 数组做同样的事情。在下一节中,我们将看看该协议有助于利用的潜在性能优势。
请注意,本文描述的功能仍在实验阶段,有些仍在开发和审查中。有关 __array_function__ 实际进展的更详细讨论,请参阅问题部分。
性能
在进一步深入之前,假设本文所述的所有性能结果都使用了以下硬件
- CPU: 6核 (12线程) Intel Core i7-7800X @ 3.50GHz
- 主内存: 16 GB
- GPU: NVIDIA Quadro GV100
- OpenBLAS 0.2.18
- cuBLAS 9.2.174
- cuSOLVER 9.2.148
现在我们来看一个例子,看看在使用 CuPy 作为后端时,__array_function__ 协议与 Dask 结合可能带来的性能优势。首先,我们创建所有稍后用于计算 SVD 的数组。请注意,我在这里的重点是 Dask 如何利用计算性能,因此在此示例中,我忽略了在 CPU 和 GPU 之间创建或复制数组所花费的时间。
import numpy as np
import cupy
import dask.array as da
x = np.random.random((10000, 1000))
y = cupy.array(x)
dx = da.from_array(x, chunks=(5000, 1000))
dy = da.from_array(y, chunks=(5000, 1000), asarray=False)
如上所示,我们有四个数组
x: 主内存中的 NumPy 数组;y: GPU 内存中的 CuPy 数组;dx: 包装在 Dask 数组中的 NumPy 数组;dy: 包装在 Dask 数组中的 CuPy 数组的副本; 将 CuPy 数组作为视图 (asarray=True) 包装在 Dask 数组中尚不支持。
在 NumPy 数组上计算 SVD
然后我们可以开始使用 NumPy 计算 x 的 SVD,因此,它在 CPU 上以单线程处理
u, s, v = np.linalg.svd(x)
之后我获得的计时信息如下
CPU times: user 3min 10s, sys: 347 ms, total: 3min 11s
Wall time: 3min 11s
超过 3 分钟似乎有点太慢了,所以现在的问题是:我们可以做得更好吗,更重要的是,无需更改我们的整个代码?
这个问题的答案是:是的,我们可以。
现在我们看看其他结果。
在包装在 Dask 数组中的 NumPy 数组上计算 SVD
首先,这是在引入 __array_function__ 协议之前您必须做的事情
u, s, v = da.linalg.svd(dx)
u, s, v = dask.compute(u, s, v)
上面的代码可能对许多项目来说非常具有限制性,因为除了传递正确的数组之外,还需要调用适当的库调度器。换句话说,需要找到代码中所有的 NumPy 调用,并根据输入的数组类型,将其替换为正确库的函数调用。在有了 __array_function__ 之后,可以使用 Dask 数组 dx 作为输入来调用相同的 NumPy 函数
u, s, v = np.linalg.svd(dx)
u, s, v = dask.compute(u, s, v)
注意: Dask 会延迟计算结果,直到被使用时才会执行,因此我们需要对结果数组调用 dask.compute() 函数来实际计算它们。
现在我们看看计时信息
CPU times: user 1min 23s, sys: 460 ms, total: 1min 23s
Wall time: 1min 13s
现在,除了将 NumPy 数组包装成 Dask 数组之外,无需更改任何代码,我们就可以看到 2 倍的加速。还不错。但是让我们回到之前的问题:我们可以做得更好吗?
在 CuPy 数组上计算 SVD
现在我们可以像对 Dask 数组一样操作,只需在 CuPy 数组 y 上简单地调用 NumPy 的 SVD 函数
u, s, v = np.linalg.svd(y)
现在我们获得的计时信息如下
CPU times: user 17.3 s, sys: 1.81 s, total: 19.1 s
Wall time: 19.1 s
现在我们看到了 4-5 倍的加速,内部调用没有任何变化!这正是我们期望利用 __array_function__ 协议带来的好处,免费加速现有代码!
让我们最后一次回到最初的问题:我们可以做得更好吗?
在包装在 Dask 数组中的 CuPy 数组上计算 SVD
现在我们可以利用 Dask 数据块分割以及 CuPy GPU 实现的优势,尽量让 GPU 保持忙碌,这仍然像这样简单
u, s, v = np.linalg.svd(dy)
u, s, v = dask.compute(u, s, v)
由此我们获得以下计时
CPU times: user 8.97 s, sys: 653 ms, total: 9.62 s
Wall time: 9.45 s
这比单线程 CuPy SVD 计算又带来了 2 倍的加速。
总而言之,我们从超过 3 分钟开始,现在通过简单地将工作分派到不同的数组上,已经缩短到不到 10 秒。
应用
现在我们将稍微谈谈 __array_function__ 协议的潜在应用。为此,我们将讨论 Dask-GLM 库,该库用于在大数据集上拟合广义线性模型。它构建在 Dask 之上,并提供了与 scikit-learn 兼容的 API。
在引入 __array_function__ 协议之前,我们需要为我们希望用作后端的每个类似 NumPy 的库重写其大部分内部实现,因此,我们需要为 Dask、CuPy 和 Sparse 各自专门实现。现在,由于这些库通过兼容的接口共享所有功能,我们完全无需更改实现,只需传入不同类型的数组作为输入,就这么简单。
scikit-learn 示例
为了展示我们获得的能力,我们来看一个 scikit-learn 示例(基于此处的原始示例)
import numpy as np
import matplotlib.pyplot as plt
from sklearn.linear_model import LinearRegression
N = 1000
# x from 0 to N
x = N * np.random.random((40000, 1))
# y = a*x + b with noise
y = 0.5 * x + 1.0 + np.random.normal(size=x.shape)
# create a linear regression model
est = LinearRegression()
然后我们可以拟合模型,
est.fit(x, y)
并获取其时间测量结果
CPU times: user 3.4 ms, sys: 0 ns, total: 3.4 ms
Wall time: 2.3 ms
然后我们可以用它对一些测试数据进行预测,
# predict y from the data
x_new = np.linspace(0, N, 100)
y_new = est.predict(x_new[:, np.newaxis])
并检查其时间测量结果
CPU times: user 1.16 ms, sys: 680 µs, total: 1.84 ms
Wall time: 1.58 ms
最后绘制结果
# plot the results
plt.figure(figsize=(4, 3))
ax = plt.axes()
ax.scatter(x, y, linewidth=3)
ax.plot(x_new, y_new, color='black')
ax.set_facecolor((1.0, 1.0, 0.42))
ax.set_xlabel('x')
ax.set_ylabel('y')
ax.axis('tight')
plt.show()
Dask-GLM 示例
我们只需要更改之前的代码中的第一个块,即导入库和创建数组的部分
import numpy as np
from dask_glm.estimators import LinearRegression
import matplotlib.pyplot as plt
N = 1000
# x from 0 to N
x = N * np.random.random((40000, 1))
# y = a*x + b with noise
y = 0.5 * x + 1.0 + np.random.normal(size=x.shape)
# create a linear regression model
est = LinearRegression(solver='lbfgs')
其余代码和绘图与之前的 scikit-learn 示例类似,为简洁起见在此省略。另请注意,我们可以调用 LinearRegression() 而不带任何参数,但为了这个示例,我们选择了收敛速度相当快的 lbfgs 求解器。
我们还可以看一下 Dask-GLM 的拟合时间结果,接着是预测时间结果
# Fitting
CPU times: user 9.66 ms, sys: 116 µs, total: 9.78 ms
Wall time: 8.94 ms
# Predicting
CPU times: user 130 µs, sys: 327 µs, total: 457 µs
Wall time: 1.06 ms
如果我们要改用 CuPy 进行计算,只需更改 3 行代码:将导入 numpy 改为导入 cupy,以及创建随机数组的两行,将其中的 np.random 替换为 cupy.random。该代码块应如下所示
import cupy
from dask_glm.estimators import LinearRegression
import matplotlib.pyplot as plt
N = 1000
# x from 0 to N
x = N * cupy.random.random((40000, 1))
# y = a*x + b with noise
y = 0.5 * x + 1.0 + cupy.random.normal(size=x.shape)
# create a linear regression model
est = LinearRegression(solver='lbfgs')
在这种情况下我们获得的计时结果是
# Fitting
CPU times: user 151 ms, sys: 40.7 ms, total: 191 ms
Wall time: 190 ms
# Predicting
CPU times: user 1.91 ms, sys: 778 µs, total: 2.69 ms
Wall time: 1.37 ms
对于本文选择的简单示例,scikit-learn 在使用 NumPy 和 CuPy 数组时都优于 Dask-GLM。可能有多种原因促成这一点,虽然我们没有深入探讨理解确切的原因及其程度,但我们可以列举一些可能的因素
- scikit-learn 可能使用了收敛速度更快的求解器;
- Dask-GLM 完全构建在 Dask 之上,而 scikit-learn 内部可能经过了高度优化;
- 对于使用 CuPy 的小型数据集,可能会发生过多的同步步骤和数据传输。
不同 Dask-GLM 求解器的性能
为了验证使用 NumPy 数组的 Dask-GLM 与 CuPy 数组相比如何,我们还对 Dask-GLM 求解器进行了一些逻辑回归基准测试。以下结果来自一个训练数据集,该数据集包含 100 维的 102、103、...、106 个特征,以及相同数量的测试特征。
注意: 我们有意省略 Dask 数组的结果,因为我们发现了一个可能导致 Dask 数组不收敛的潜在错误。
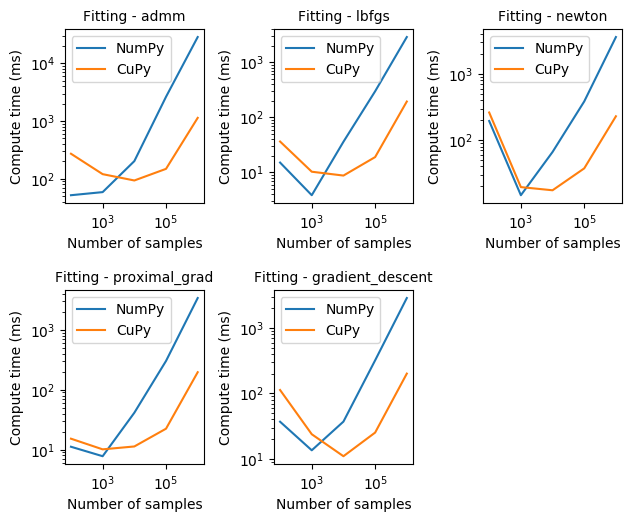
从上面的图表中观察到的结果可以看出,对于使用任何 Dask-GLM 求解器进行拟合,CuPy 可以比 NumPy 快一个数量级。另请注意,为了更容易可视化,两个坐标轴都以对数刻度表示。
另一个有趣的现象是,对于少量样本,收敛可能需要更长时间。然而,正如我们通常所希望的,收敛所需的计算时间与样本数量呈线性关系。
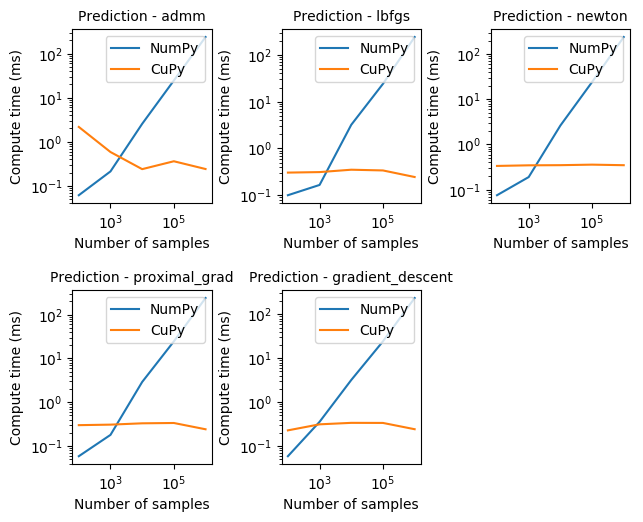
如上所示,使用 CuPy 进行预测可以比 NumPy 成比例地快得多,对于所有求解器基本保持恒定,并且快约 3-4 个数量级。
问题
在本节中,我们将描述自 2019 年 2 月以来已经完成和仍在进行的工作,以实现前面章节中描述的功能。如果您对所有细节不感兴趣,可以完全跳过本节。
已解决的问题
自 2019 年 2 月初以来,在不同项目中深入支持 __array_function__ 协议方面取得了实质性进展,这一趋势仍在继续,并将持续到 3 月份。下面列出了已解决或正在审查中的问题
__array_function__协议依赖项已在 CuPy PR #2029 中修复;- 使用 CuPy 后端时 Dask 的 mean() 和 moment() 问题 Dask Issue #4481,已在 Dask PR #4513 和 Dask PR #4519 中修复;
- 替换 SciPy 中在 CuPy 等库中可能不可用的 NumPy 函数别名,已在 SciPy PR #9888 中修复;
- 允许创建任意形状的数组,使用输入数组作为要创建的新数组的参考,正在 NumPy PR #13043 中审查;
- 使用 CuPy 的多线程问题,首次在 Dask Issue #4487、CuPy Issue #2045 和 CuPy Issue #1109 中发现,目前正在 CuPy PR #2053 中审查;
- 在 CuPy 数组上调用 Dask 的
flatnonzero()时缺少cupy.compress(),首次在 Dask Issue #4497 中发现,正在 Dask PR #4548 中审查, - Dask 对
__array_function__的支持,正在 Dask PR #4567 中审查。
已知问题
目前,我们正在解决的最大问题之一是与 Dask issue #4490 相关,该问题在我们首次在 CuPy 数组上调用 Dask 的 diag() 时发现。这需要在 Dask 的 Array 类上进行一些更改,并在 Dask 代码库的大部分区域进行后续更改。我不会在这里详细说明,但我们解决这个问题的方法是在 Dask Array 中添加一个新的属性 _meta,以替代目前存在的简单 dtype。这个新属性不仅将包含 dtype 信息,还将包含用于创建 Array 的后端类型的空数组,从而使我们能够在内部重新构造后端类型的数组,而无需明确知道它是 NumPy、CuPy、Sparse 还是任何其他类似 NumPy 的数组。更多详细信息,请参阅 Dask Issue #2977。
我们还发现了一些正在讨论中的问题
- 将 Sparse 用作 Dask 后端,在 Dask Issue #4523 中讨论;
- 在 CuPy 数组上调用 Dask 的
fix()依赖于__array_wrap__,在 Dask Issue #4496 和 CuPy Issue #589 中讨论; - 允许强制转换
__array_function__,在 NumPy Issue #12974 中讨论。
未来工作
有多种可能性可以提供更丰富的 Dask 体验,其中一些在短期和中期可能非常有趣
-
将 Dask-cuDF 与 Dask-GLM 结合使用,展示整个生态系统中有趣的实际应用;
-
更多全面的 Dask-GLM 示例和基准测试;
-
在 Dask-GLM 中支持更多模型;
-
深入研究 Dask-GLM 与 scikit-learn 的性能对比;
-
分析 CuPy 矩阵-矩阵乘法操作 (GEMM) 的性能,并将其与分布式 Dask 操作的矩阵-向量乘法操作 (GEMV) 进行比较。
博客评论由 Disqus 提供支持