使用 Dask Array 加载大型图像数据
作者:John Kirkham
内容提要
本文探讨了使用 Dask array 加载大型图像数据栈的简单工作流程。
特别是,我们首先从一个包含 TIFF 文件的目录开始,文件中的图像示例如下
$ $ ls raw/ | head
ex6-2_CamA_ch1_CAM1_stack0000_560nm_0000000msec_0001291795msecAbs_000x_000y_000z_0000t.tif
ex6-2_CamA_ch1_CAM1_stack0001_560nm_0043748msec_0001335543msecAbs_000x_000y_000z_0000t.tif
ex6-2_CamA_ch1_CAM1_stack0002_560nm_0087497msec_0001379292msecAbs_000x_000y_000z_0000t.tif
ex6-2_CamA_ch1_CAM1_stack0003_560nm_0131245msec_0001423040msecAbs_000x_000y_000z_0000t.tif
ex6-2_CamA_ch1_CAM1_stack0004_560nm_0174993msec_0001466788msecAbs_000x_000y_000z_0000t.tif
并展示了如何使用dask-image库将这些文件拼接成大型的惰性(lazy)数组
>>> import dask_image
>>> x = dask_image.imread.imread('raw/*.tif')
或者通过编写自己的 Dask delayed 图像读取函数。
|
总有一天,我们将能够对这个 dask 数组执行复杂的计算。
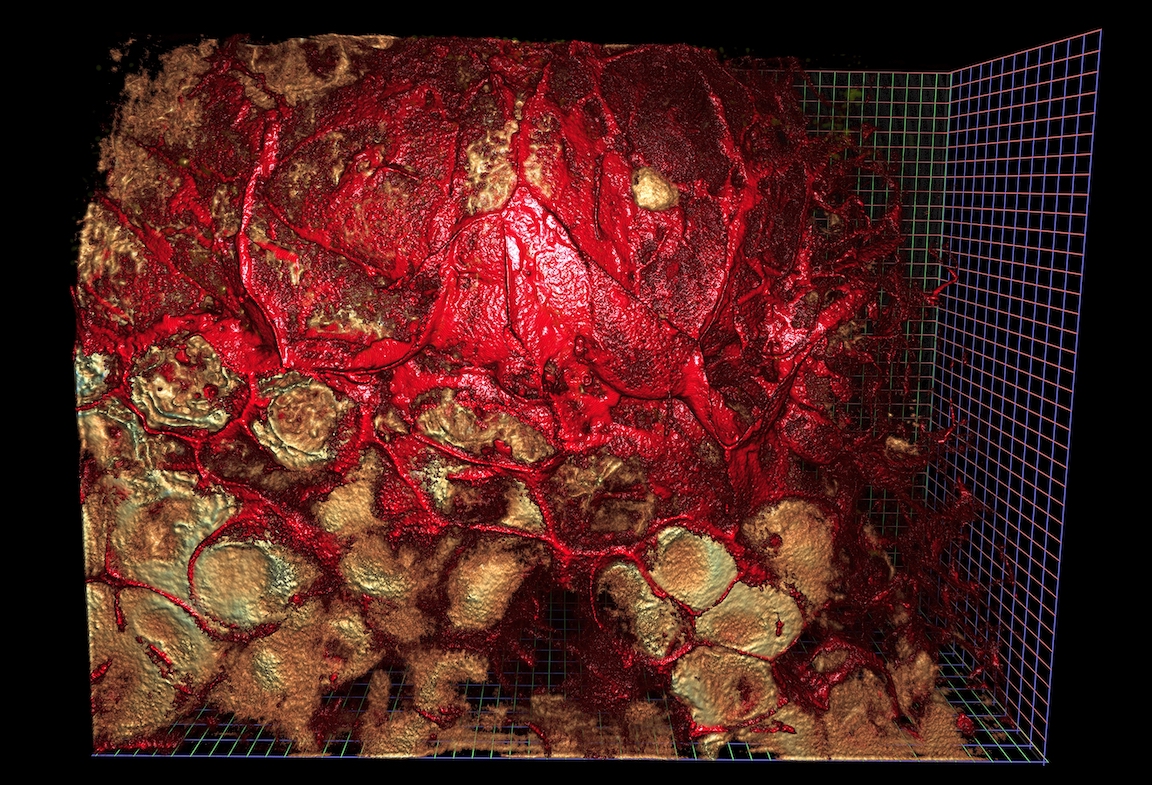
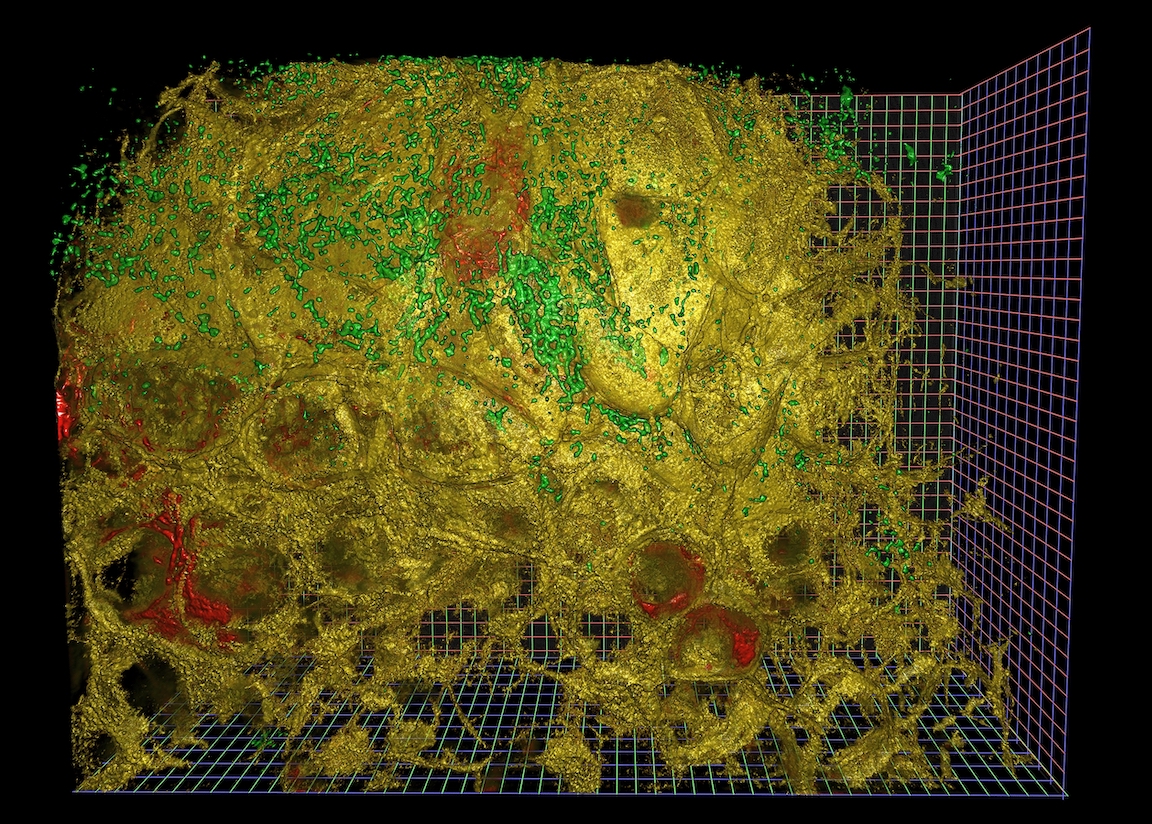
免责声明:本文中不会生成如上所示的渲染图像。这些图像是使用 NVidia IndeX 创建的,这是一个与本文讨论内容完全独立的工具链。本文仅涵盖图像加载的第一步。
系列概述
在采集大量成像数据的领域中,一个常见的情况是将较小的采集数据写入许多小文件。这些文件可以平铺一个更大的空间,从更长的时间段中进行子采样,并且可能包含多个通道。采集技术本身通常是尖端的,并在视野范围、分辨率和质量方面不断突破。
一旦采集到这些数据,就会带来一系列挑战。通常设计和测试用于处理少量数据的算法需要扩展以处理完整的数据集。一开始可能不清楚什么方法会实际奏效,因此探索仍然是整个过程的非常重要的一部分。
从历史上看,这种分析过程涉及大量定制代码。通常,分析过程是通过一系列脚本拼接而成的,这些脚本可能使用多种不同的语言,并将各种中间结果写入磁盘。得益于现代工具的进步,这些过程可以得到显著改进。在本系列博客文章中,我们将概述图像科学家如何利用不同的工具,朝着构建高级、友好、连贯、交互式的分析管道迈进。
文章概述
本文特别侧重于如何从 Python 中并行加载和管理大型图像数据栈。
加载大型图像数据可能是一个复杂且通常独特的问题。不同的团队可能会选择将其存储在磁盘上的许多文件中、使用现成的或定制的数据库解决方案,或者选择将其存储在云端。出于各种原因,同一团队内的所有数据集的处理方式可能不同。简而言之,这意味着数据加载是一个困难且昂贵的问题。
尽管数据存储方式多种多样,但各团队通常希望将相同的分析管道重新应用于这些数据集。然而,如果数据管道与特定的数据加载方式紧密耦合,那么在后续分析步骤中重用现有管道可能会变得非常困难,甚至不可能。换句话说,加载和分析步骤之间存在阻碍,这使得实现可重用性变得困难。
拥有模块化和通用的数据加载方式,可以轻松地以标准方式呈现不同存储格式的数据。此外,将数据以标准方式提供给分析管道,可以使管道的分析部分专注于其最擅长的分析工作!总的来说,这应该能够解耦这两个组件,从而改善管道所有环节中用户的体验。
我们将使用由加州大学伯克利分校高级生物成像中心的Gokul Upadhyayula慷慨提供的图像数据,这些数据在这篇论文(预印本)中有所讨论,尽管本文介绍的工作流程适用于任何类型的成像数据或通常的数组数据。
使用 Dask 加载图像数据
让我们再次从文章开头提到的图像数据开始
$ $ ls /path/to/files/raw/ | head
ex6-2_CamA_ch1_CAM1_stack0000_560nm_0000000msec_0001291795msecAbs_000x_000y_000z_0000t.tif
ex6-2_CamA_ch1_CAM1_stack0001_560nm_0043748msec_0001335543msecAbs_000x_000y_000z_0000t.tif
ex6-2_CamA_ch1_CAM1_stack0002_560nm_0087497msec_0001379292msecAbs_000x_000y_000z_0000t.tif
ex6-2_CamA_ch1_CAM1_stack0003_560nm_0131245msec_0001423040msecAbs_000x_000y_000z_0000t.tif
ex6-2_CamA_ch1_CAM1_stack0004_560nm_0174993msec_0001466788msecAbs_000x_000y_000z_0000t.tif
使用 Scikit-Image 加载单个示例图像
为了加载单个图像,我们使用Scikit-Image
>>> import glob
>>> filenames = glob.glob("/path/to/files/raw/*.tif")
>>> len(filenames)
597
>>> import imageio
>>> sample = imageio.imread(filenames[0])
>>> sample.shape
(201, 1024, 768)
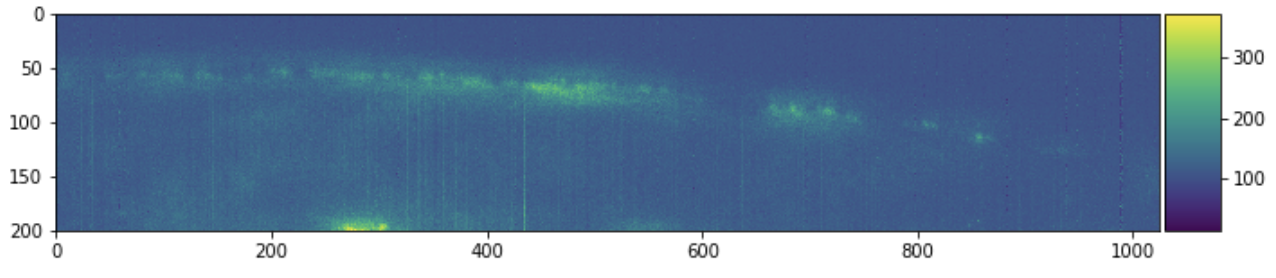
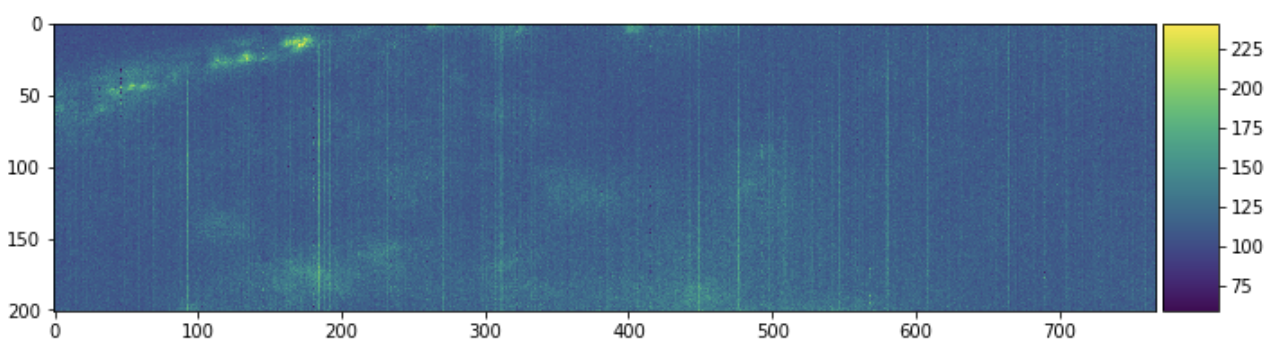
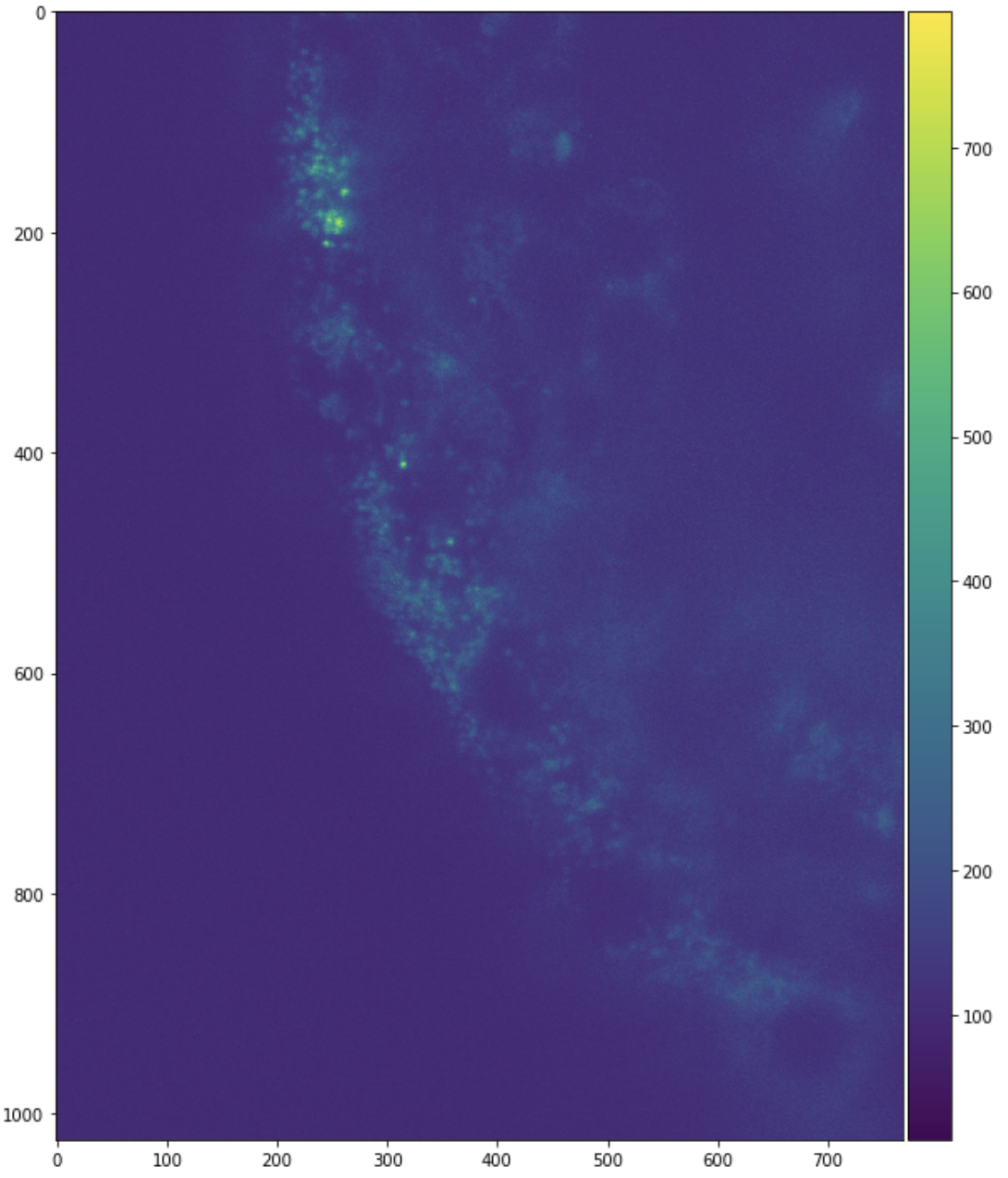
每个文件名对应于一个更大图像的某个 3D 数据块。我们可以查看这个单一 3D 数据块的几个 2D 切片,以获取一些背景信息。
import matplotlib.pyplot as plt
import skimage.io
plt.figure(figsize=(10, 10))
skimage.io.imshow(sample[:, :, 0])
plt.figure(figsize=(10, 10))
skimage.io.imshow(sample[:, 0, :])
plt.figure(figsize=(10, 10))
skimage.io.imshow(sample[0, :, :])
探究文件名结构
这些切片仅来自一个更大聚合图像中的一个数据块。我们在这里的兴趣是将这些部分组合成一个大型图像栈。文件名中常见一种命名结构。每个文件名可能指示一个通道、时间步长和空间位置,其中<i>是一些数值(可能带有单位)。单个文件名可能包含更多或更少的信息,并且其标记方式可能与我们的不同。
mydata_ch<i>_<j>t_<k>x_<l>y_<m>z.tif
原则上,使用 NumPy 我们可以分配一个巨大的数组,然后迭代加载图像并将其放入这个巨大的数组中。
full_array = np.empty((..., ..., ..., ..., ...), dtype=sample.dtype)
for fn in filenames:
img = imageio.imread(fn)
index = get_location_from_filename(fn) # We need to write this function
full_array[index, :, :, :] = img
然而,如果我们的数据很大,我们就无法像这样一次性将所有数据加载到单个 Numpy 数组中,而是需要更巧妙地高效处理。这里的一种方法是使用Dask,它可以轻松处理超出内存的工作负载。
使用 Dask Array 惰性(Lazy)加载图像
现在我们学习如何使用 Dask array 惰性加载和拼接图像数据。我们将从简单的例子开始,然后转向使用这个更复杂数据集的完整例子。
我们可以使用Dask Delayed延迟imageio.imread的调用。
import dask
import dask.array as da
lazy_arrays = [dask.delayed(imageio.imread)(fn) for fn in filenames]
lazy_arrays = [da.from_delayed(x, shape=sample.shape, dtype=sample.dtype)
for x in lazy_arrays]
注意:这里我们假设所有图像都具有与上面加载的示例文件相同的形状(shape)和数据类型(dtype)。并非总是如此。请参阅下方“未来工作”部分中关于dask_image的说明,了解替代方案。
我们尚未将这些数据拼接在一起。我们有数百个单数据块的 Dask 数组,每个数组都从磁盘惰性加载一个单一的 3D 数据块。让我们看一个单一的数组。
>>> lazy_arrays[0]
|
这是一个由一个单一 300MB 数据块组成的惰性 3 维 Dask 数组。该数据块是通过加载一个特定的 TIFF 文件创建的。通常,Dask 数组由许多数据块组成。我们可以使用诸如 da.concatenate 和 da.stack 等函数将许多这样的单数据块 Dask 数组连接成一个多数据块 Dask 数组。
在这里,我们将前十个 Dask 数组沿着几个轴连接起来,以便更容易理解其外观。请同时注意左侧表格和右侧图像中,随着我们改变axis=参数,形状如何变化。
da.concatenate(lazy_arrays[:10], axis=0)
|
da.concatenate(lazy_arrays[:10], axis=1)
|
da.concatenate(lazy_arrays[:10], axis=2)
|
或者,如果想创建一个新维度,我们会使用da.stack。在这种情况下请注意,我们已经用尽了容易可视化的维度,因此您应该更多地关注左侧表格输入中列出的形状,而不是右侧的图片。请注意,我们将这些 3D 图像堆叠成了 4D 图像。
da.stack(lazy_arrays[:10])
|
这些是常见的情况,即您有一个单一的轴,沿着该轴您希望将图像拼接在一起。
完整示例
这对于沿单一轴组合效果很好。但是,如果需要跨多个轴组合,则需要执行多个连接步骤。幸运的是,有一个更简单的选项da.block,如果您提供嵌套的 dask 数组列表,它可以一次沿多个轴进行连接。
a = da.block([[laxy_array_00, lazy_array_01],
[lazy_array_10, lazy_array_11]])
我们现在执行以下操作
- 解析每个文件名,了解它在大型数组中应位于何处
- 查看每个相关维度中有多少文件
- 分配一个适当大小的 NumPy object-dtype 数组,该数组的每个元素将 holding 一个单数据块的 Dask 数组
- 遍历文件名,将相应的 Dask 数组插入到正确的位置
- 对结果调用
da.block
这段代码有点复杂,但展示了它在实际环境中的样子
# Get various dimensions
fn_comp_sets = dict()
for fn in filenames:
for i, comp in enumerate(os.path.splitext(fn)[0].split("_")):
fn_comp_sets.setdefault(i, set())
fn_comp_sets[i].add(comp)
fn_comp_sets = list(map(sorted, fn_comp_sets.values()))
remap_comps = [
dict(map(reversed, enumerate(fn_comp_sets[2]))),
dict(map(reversed, enumerate(fn_comp_sets[4])))
]
# Create an empty object array to organize each chunk that loads a TIFF
a = np.empty(tuple(map(len, remap_comps)) + (1, 1, 1), dtype=object)
for fn, x in zip(filenames, lazy_arrays):
channel = int(fn[fn.index("_ch") + 3:].split("_")[0])
stack = int(fn[fn.index("_stack") + 6:].split("_")[0])
a[channel, stack, 0, 0, 0] = x
# Stitch together the many blocks into a single array
a = da.block(a.tolist())
|
这是一个 180 GB 的逻辑数组,由大约 600 个块组成,每个块大小为 300 MB。现在我们可以使用Dask Array 对这个数组进行类似 NumPy 的常规计算,但这将留到以后的文章中介绍。
>>> # array computations would work fine, and would run in low memory
>>> # but we'll save actual computation for future posts
>>> a.sum().compute()
保存数据
为了简化未来的数据加载,我们使用to_zarr方法将其存储在像Zarr这样的大型分块数组格式中。
a.to_zarr("mydata.zarr")
我们可以添加关于图像数据的额外信息作为属性。这既使未来用户更容易(他们可以使用da.from_zarr一行代码读取整个数据集),又大大提高了性能,因为 Zarr 是一种为计算高效编码的分析就绪格式。
Zarr 默认使用Blosc库进行压缩。对于科学成像数据,我们可以选择传递压缩选项,以在压缩率和速度之间取得良好的权衡,并优化压缩性能。
from numcodecs import Blosc
a.to_zarr("mydata.zarr", compressor=Blosc(cname='zstd', clevel=3, shuffle=Blosc.BITSHUFFLE))
未来工作
上述工作流程通用且直接。它在简单情况下工作良好,并且也能很好地扩展到更复杂的情况,前提是您愿意围绕自定义逻辑编写一些循环和解析代码。它既可以在单台小型笔记本电脑上运行,也可以在大型 HPC 或云集群上运行。如果您有一个函数可以将文件名转换为 NumPy 数组,您就可以使用该函数、Dask Delayed和Dask Array生成大型的惰性 Dask 数组。
Dask Image
然而,如果我们稍微专业化一下,可以为用户提供便利。例如,Dask Image 库提供了一个并行图像读取函数,它在简单情况下自动化了我们上述的大部分工作。
>>> import dask_image
>>> x = dask_image.imread.imread('raw/*.tif')
类似地,像Xarray这样的库提供了其他文件格式的读取器,例如 GeoTIFF。
随着各个领域越来越多地进行我们上面所做的工作,他们倾向于将常见模式编写成领域特定的库,这进一步提高了这些工具的可访问性和用户群。
GPU
如果我们有一些闲置的特殊硬件,例如几个 GPU,我们可以将数据转移到上面,并使用像 CuPy 这样的库进行计算,CuPy 与 NumPy 非常相似。因此,可以受益于上面列出的相同操作,但附加了 GPU 带来的性能提升。
import cupy as cp
a_gpu = a.map_blocks(cp.asarray)
计算
最后,在未来的博客文章中,我们计划讨论如何使用常见的图像处理工作负载(如重叠模板函数、分割和反卷积)以及与其他库(如 ITK)集成,对大型 Dask 数组进行计算。
博客评论由 Disqus 提供支持