图像分析再探 Dask + CuPy + RL
作者:John Kirkham (NVIDIA) 和 Ben Zaitlen (NVIDIA)
摘要
去年我们实验了 Dask/ITK/Scikit-Image 来对三维图像栈进行大规模图像分析。具体来说,我们研究了反卷积(deconvolution),这是一种常见的图像去模糊(deblur)方法。一年后的现在,我们带着对 Dask 和 CuPy 如何交互的更好理解、增强的序列化方法以及来自开源社区的支持,回到了这些实验。本文将探讨以下内容:
- 为 CPU + GPU 实现一种常见的反卷积方法
- 利用 Dask 对大型数据集执行反卷积
- 使用 Napari 图像查看器探索结果
图像分析再探
之前我们使用了 ITK 和 Scikit-Image 中的 Richardson Lucy (RL) 反卷积算法。我们当时暂停了,去理论探讨 GPU 如何潜在地帮助加速这些工作流程。从 Scikit-Image 的实现开始,我们天真地尝试将 scipy.signal.convolve 调用替换为 cupyx.scipy.ndimage.convolve,虽然性能有所提升,但并未显著提升——也就是说,我们没有达到我们期望的 100 倍速度。
正如数学中经常出现的情况一样,一个在某种表示形式下解决起来效率不高的问题,往往可以通过事先对数据进行转换来提高效率。在这种新的表示形式下,我们可以更容易地解决相同的问题(在本例中是卷积),然后再将结果转换回更熟悉的表示形式。对于卷积来说,我们应用的转换叫做 快速傅里叶变换(FFT)。一旦应用了它,我们就可以使用简单的乘法来对数据进行卷积。
事实证明,FFT 转换在 CPU 和 GPU 上都非常快。类似地,我们可以用 FFT 编写的算法也得到了加速。这是图像处理领域中用于加速卷积的一种常用技术。尽管增加了进行 FFT 的步骤,但转换成本加上算法成本仍然低于在实空间中执行原始算法的成本。我们(以及我们之前的其他人)发现,对于 Richardson Lucy(在 CPU 和 GPU 上)来说,情况确实如此,当我们使用 Dask 在多个 GPU 上进行并行化时,性能持续提升。
来自开源社区的帮助
基于 FFT 的 RL 等效算法已经存在一段时间了,来自 太阳动力学天文台 的优秀工程师们构建并分享了一个 NumPy/CuPy 实现,作为 大气成像组件 Python 包 (aiapy) 的一部分。我们稍微修改了他们的实现,使其能够处理 3D 和 2D 点扩散函数,并利用 NEP-18 方便地将 NumPy 和 CuPy 分派给相应的函数。
def deconvolve(img, psf=None, iterations=20):
# Pad PSF with zeros to match image shape
pad_l, pad_r = np.divmod(np.array(img.shape) - np.array(psf.shape), 2)
pad_r += pad_l
psf = np.pad(psf, tuple(zip(pad_l, pad_r)), 'constant', constant_values=0)
# Recenter PSF at the origin
# Needed to ensure PSF doesn't introduce an offset when
# convolving with image
for i in range(psf.ndim):
psf = np.roll(psf, psf.shape[i] // 2, axis=i)
# Convolution requires FFT of the PSF
psf = np.fft.rfftn(psf)
# Perform deconvolution in-place on a copy of the image
# (avoids changing the original)
img_decon = np.copy(img)
for _ in range(iterations):
ratio = img / np.fft.irfftn(np.fft.rfftn(img_decon) * psf)
img_decon *= np.fft.irfftn((np.fft.rfftn(ratio).conj() * psf).conj())
return img_decon
对于一个 1.3 GB 的图像,我们测量结果如下:
- CuPy:20 次迭代约 3 秒
- NumPy:2 次迭代约 36 秒
我们看到,以 10 倍的迭代次数获得了 10 倍的速度提升——非常接近我们期望的 100 倍加速!接下来,让我们看看这个实现在真实的生物数据和 Dask 下的表现如何……
定义 Dask 集群并加载数据
NIH 的 Shroff 教授实验室提供了样本数据。这些数据最初是 3D TIFF 文件,我们后来将其转换为 Zarr 格式,形状为 (950, 2048, 2048)。
我们首先在 DGX2(单个节点包含 16 个 GPU)上创建一个 Dask 集群,并加载存储在 Zarr 中的图像
from dask.distributed import Client
from dask_cuda import LocalCUDACluster
import dask.array as da
import rmm
import cupy as cp
cluster = LocalCUDACluster(
local_directory="/tmp/bzaitlen",
enable_nvlink=True,
rmm_pool_size="26GB",
)
client = Client(cluster)
client.run(
cp.cuda.set_allocator,
rmm.rmm_cupy_allocator
)
imgs = da.from_zarr("/public/NVMICROSCOPY/y1z1_C1_A.zarr/")
|
从上面的 Dask 输出可以看出,数据是 950 张图像组成的 Z 堆栈,每个切片的大小为 2048x2048。对于这个数据集,如果我们处理更大的块,可以提升 GPU 性能。此外,我们需要确保块的大小至少与 PSF(在本例中是 (128, 128, 128))一样大。由于我们在拥有 16 个 GPU 的 DGX2 上进行工作,如果我们适当地对数据进行 rechunk,就可以轻松地将数据放入并在每个 GPU 上执行反卷积。
# chunk with respect to PSF shape (128, 128, 128)
imgs = imgs.rechunk(chunks={0: 190, 1: 512, 2: 512})
imgs
| 数组 | 块 | |
|---|---|---|
| 字节 | 7.97 GB | 99.61 MB |
| 形状 | (950, 2048, 2048) | (190, 512, 512) |
| 计数 | 967 个任务 | 80 个块 |
| 类型 | uint16 | numpy.ndarray |
接下来,我们将数据转换为 float32,因为数据可能不是浮点类型。此外,32 位计算速度比 64 位稍快,并节省一些内存。下面我们将 cupy.asarray 映射到每个数据块上。cupy.asarray 将数据从主机内存 (NumPy) 移动到设备/GPU (CuPy)。
imgs = imgs.astype(np.float32)
c_imgs = imgs.map_blocks(cp.asarray)
|
现在我们得到了一个由 16 个 CuPy 数据块组成的 Dask 数组。请注意 Dask 在 SVG 输出中提供了很好的类型信息。当我们从 NumPy 迁移到 CuPy 时,上面的块图显示 Type: cupy.ndarray —— 这是一个很好的健全性检查。
在运行反卷积之前,我们需要的最后一部分是 PSF,它也应该加载到 GPU 上
import skimage.io
psf = skimage.io.imread("/public/NVMICROSCOPY/PSF.tif")
c_psf = cp.asarray(psf)
最后,我们使用 map_overlap 和 deconvolve 函数对 Dask 数组进行调用
out = da.map_overlap(
deconvolve,
c_imgs,
psf=c_psf,
iterations=100,
meta=c_imgs._meta,
depth=tuple(np.array(c_psf.shape) // 2),
boundary="periodic"
)
out
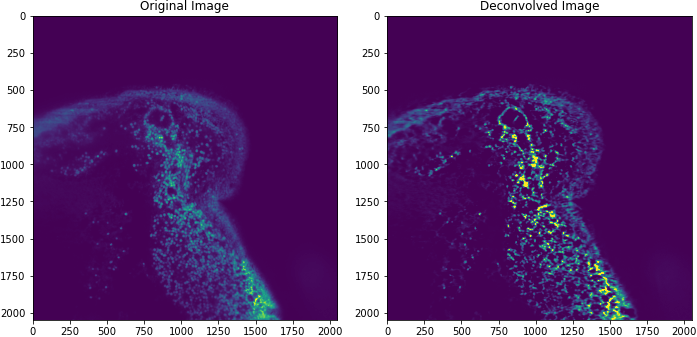
上面的图像取自小鼠肠道。
借助 Dask 和多个 GPU,我们测量到对一个 16GB 图像进行反卷积仅需约 30 秒!但这仅仅是迈向加速图像科学的第一步。
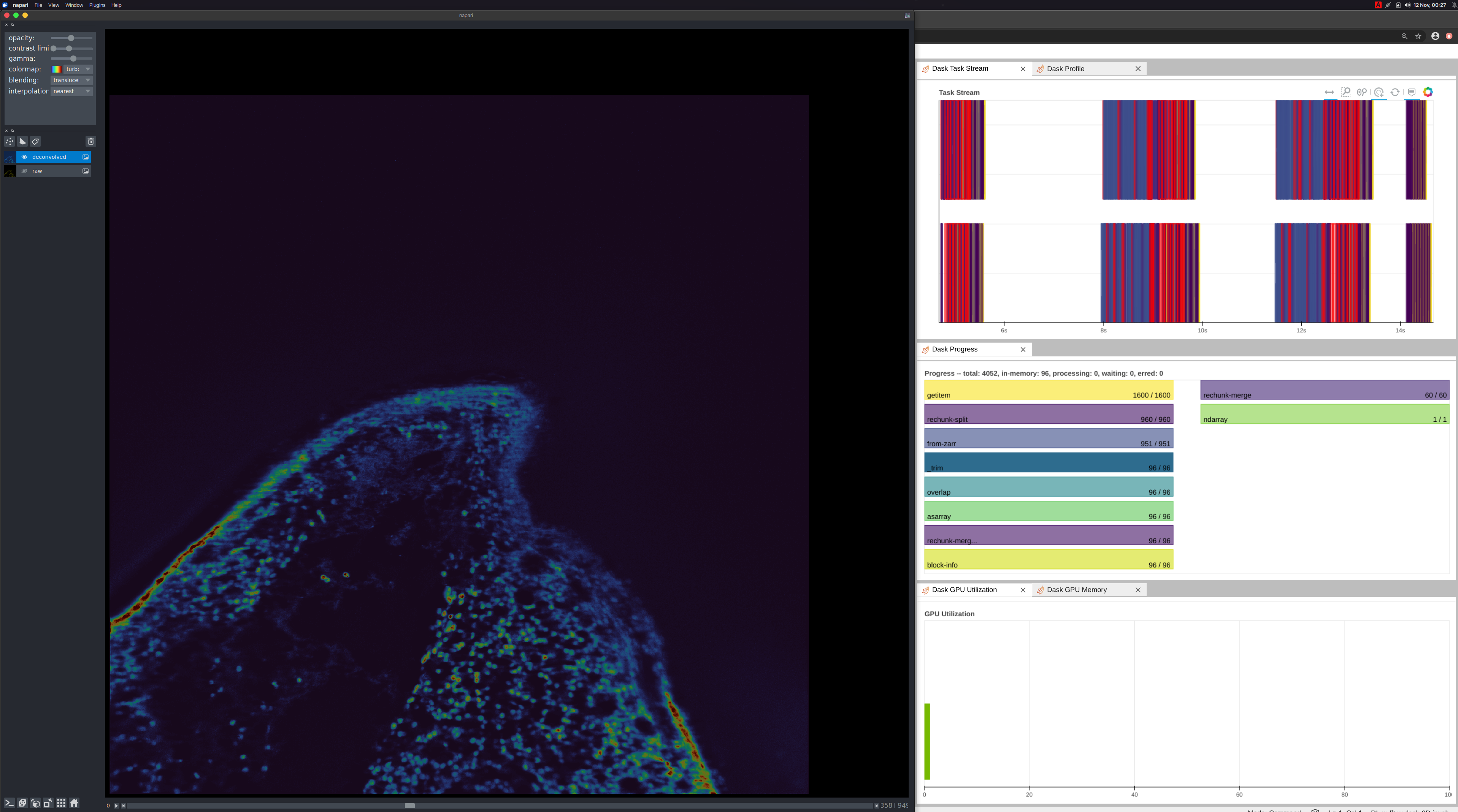
Napari
反卷积只是图像科学家或显微镜学家所需的一个操作和一种工具。在研究底层生物学时,他们还需要其他工具。在进入下一步之前,他们需要工具来可视化数据。Napari,一个在 PyData 生物生态系统中使用的多维图像查看器,是可视化这些数据的好工具。作为实验,我们在连接了 NVLink 的本地工作站上运行了相同的工作流程,该工作站配备了 2 个 Quadro RTX 8000 GPU。示例 Notebook
通过在数组中添加 map_blocks 调用,我们可以将数据从 GPU 回到 CPU(从设备回到主机)。
def cupy_to_numpy(x):
import cupy as cp
return cp.asnumpy(x)
np_out = out.map_blocks(cupy_to_numpy, meta=out)
当用户在 Napari UI 上移动滑块时,我们指示 Dask 执行以下操作:
- 将数据从磁盘加载到 GPU (CuPy)
- 计算反卷积
- 移回主机 (NumPy)
- 使用 Napari 渲染
这大约有一秒的延迟,对于一个简单的实现来说已经很不错了!我们可以通过添加缓存、使用 map_overlap 改进通信以及优化反卷积核来进一步改进。
结论
现在我们展示了如何使用 Dask + CuPy 执行 Richardson-Lucy 反卷积。这只需要最少量的代码。将其与图像查看器 (Napari) 结合使用,我们就能够检查数据和我们的结果。通过整合 PyData 库:Dask、CuPy、Zarr 和 Napari,并结合新的反卷积核,所有这些都表现得相当好。希望这能为你开始分析自己的数据提供一个好的模板,并展示自定义工作流程的丰富性和易于表达性。如果你遇到任何挑战,请在 Dask 问题跟踪器 上联系我们,我们将很乐意与你交流 :)
博客评论由 Disqus 提供