使用 Dask 进行图像分割
作者:Genevieve Buckley
执行摘要
我们将介绍如何使用 dask-image 库创建一个基本的图像分割流程。
目录
这篇博文的内容最初是 2020 年的一次会议演讲。
只看代码
如果你想自己运行,你需要从 Broad Bioimage Benchmark Collection 下载示例数据:https://bbbc.broadinstitute.org/BBBC039
并安装这些依赖
pip install dask-image>=0.4.0 tifffile
这是我们的完整流程
import numpy as np
from dask_image.imread import imread
from dask_image import ndfilters, ndmorph, ndmeasure
images = imread('data/BBBC039/images/*.tif')
smoothed = ndfilters.gaussian_filter(images, sigma=[0, 1, 1])
thresh = ndfilters.threshold_local(smoothed, blocksize=images.chunksize)
threshold_images = smoothed > thresh
structuring_element = np.array([[[0, 0, 0], [0, 0, 0], [0, 0, 0]], [[0, 1, 0], [1, 1, 1], [0, 1, 0]], [[0, 0, 0], [0, 0, 0], [0, 0, 0]]])
binary_images = ndmorph.binary_closing(threshold_image, structure=structuring_element)
label_images, num_features = ndmeasure.label(binary_image)
index = np.arange(num_features)
area = ndmeasure.area(images, label_images, index)
mean_intensity = ndmeasure.mean(images, label_images, index)
你可以继续阅读本图像分割流程的逐步讲解,或者跳到关于自定义函数、扩展计算规模或GPU 加速的部分。
图像分割流程
我们的基本图像分割流程有五个步骤
- 读取数据
- 图像过滤
- 分割对象
- 形态学操作
- 测量对象
设置 Python 环境
在开始之前,我们需要设置 Python 虚拟环境。
至少需要
pip install dask-image>=0.4.0 tifffile matplotlib
可选地,你还可以安装 napari 图像查看器来可视化图像分割结果。
pip install "napari[all]"
要在 IPython 或 jupyter 中使用 napari,在调用 napari 之前在一个单元格中运行 %gui qt magic 命令。更多详情请参阅 napari 文档。
下载示例数据
我们将使用公开可用的图像数据集 BBBC039 Caicedo et al. 2018,该数据集可在 Broad Bioimage Benchmark Collection Ljosa et al., Nature Methods, 2012 获取。你可以在这里下载数据集:https://bbbc.broadinstitute.org/BBBC039
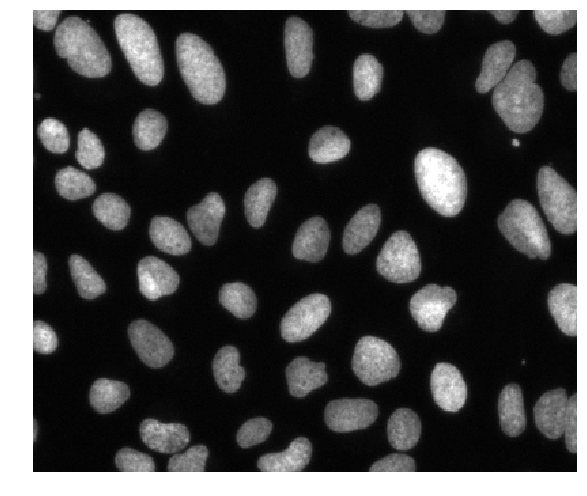
这些是荧光显微镜图像,我们可以看到单个细胞的细胞核。
步骤 1: 读取数据
我们的图像分割流程的第一步是读取图像数据。我们可以使用 dask-image imread 函数来完成。
我们传递示例数据中包含 *.tif 图像的文件夹路径。
from dask_image.imread import imread
images = imread('data/BBBC039/images/*.tif')
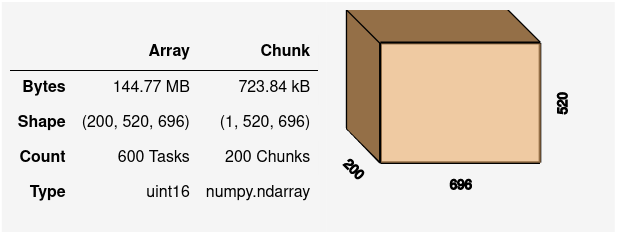
默认情况下,磁盘上的每个独立的 .tif 文件都成为了我们的 Dask 数组中的一个块(chunk)。
步骤 2: 图像过滤
对图像进行少量模糊处理进行去噪,可以改善后续的分割效果。这在许多图像分割流程中是常见的第一步。我们可以使用 dask-image 的 gaussian_filter 函数来完成。
from dask_image import ndfilters
smoothed = ndfilters.gaussian_filter(images, sigma=[0, 1, 1])
步骤 3: 分割对象
接下来,我们想将图像中的对象与背景分离。有很多不同的方法可以做到这一点。由于我们有荧光显微镜图像,我们将使用阈值分割方法。
绝对阈值
我们可以设置一个绝对阈值,低于该阈值的像素强度值将被视为背景的一部分。
absolute_threshold = smoothed > 160
让我们使用 napari 图像查看器查看这些图像。首先我们需要使用 %gui qt magic 命令
%gui qt
现在我们可以用 napari 查看图像了
import napari
viewer = napari.Viewer()
viewer.add_image(absolute_threshold)
viewer.add_image(images, contrast_limits=[0, 2000])

但这里有个问题。
当我们查看不同图像帧的结果时,很明显没有一个“放之四海而皆准”的绝对阈值可以使用。数据集中的一些图像背景相当明亮,而另一些荧光细胞核的亮度较低。我们将不得不尝试另一种阈值分割方法。
局部阈值
我们可以使用局部阈值分割方法来改善分割效果。
如果对每个图像帧独立计算阈值,那么我们可以避免由于帧间背景强度波动引起的问题。
thresh = ndfilters.threshold_local(smoothed, images.chunksize)
threshold_images = smoothed > thresh
# Let's take a look at the images with napari
viewer.add_image(threshold_images)
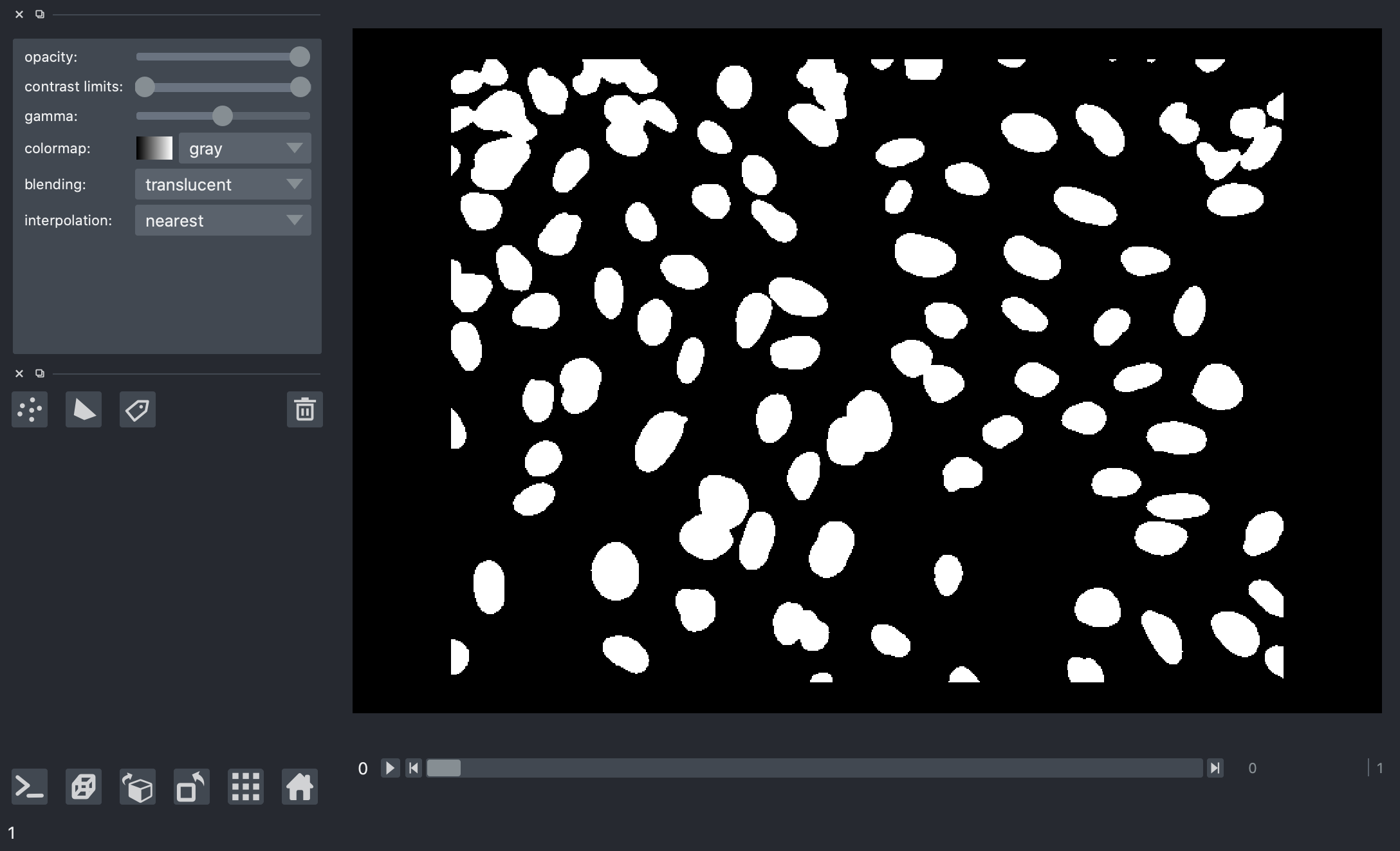
这里的结果看起来好多了,这是细胞核与背景之间更干净的分离,并且适用于所有图像帧。
步骤 4: 形态学操作
现在我们已经从阈值得到一个二值掩膜,我们可以通过一些形态学操作对其进行清理。
形态学操作是我们对二值图像中结构形状所做的改变。我们将在此简要描述一些基本概念,但更详细的参考可以查看 OpenCV 文档中的这个精彩页面。
腐蚀(Erosion)是一种操作,它会侵蚀或“吃掉”二值图像中结构的边缘。

图片来源:OpenCV 文档
膨胀(Dilation)与腐蚀相反。通过膨胀,二值图像中结构的边缘会被扩展。

图片来源:OpenCV 文档
我们可以以不同的方式组合形态学操作以获得有用的效果。
形态学开运算(morphological opening)是先进行腐蚀,然后进行膨胀的操作。

图片来源:OpenCV 文档
在上面的示例图像中,我们可以看到左侧有一个噪声斑点背景。如果用于形态学操作的结构元素大于噪声斑点的大小,它们将在第一次腐蚀步骤中完全消失。然后,当进行第二次膨胀步骤时,背景中已经没有噪音残留可供膨胀。因此,我们去除了背景中的噪音,同时我们感兴趣的主要结构(在此示例中为 J 形)几乎完美地恢复了。
让我们使用这种形态学开运算的技巧来清理我们分割流程中的二值图像。
from dask_image import ndmorph
import numpy as np
structuring_element = np.array([
[[0, 0, 0], [0, 0, 0], [0, 0, 0]],
[[0, 1, 0], [1, 1, 1], [0, 1, 0]],
[[0, 0, 0], [0, 0, 0], [0, 0, 0]]])
binary_images = ndmorph.binary_opening(threshold_images, structure=structuring_element)
你会注意到,在这里我们需要稍微注意一下结构元素(structuring element)。我们的所有图像帧都合并在一个 Dask 数组中,但我们只想对每个图像帧独立地应用形态学操作。为了做到这一点,我们在默认的 2D 结构元素的两层零之间夹入它。这意味着相邻的图像帧对结果没有影响。
# Default 2D structuring element
[[0, 1, 0],
[1, 1, 1],
[0, 1, 0]]
步骤 5: 测量对象
任何图像处理流程的最后一步都是进行某种测量。我们将把我们的二值掩膜转换为标签图像,然后测量这些对象的强度和大小。
为了让本教程中的计算时间保持快速简便,我们将只测量数据的一小部分子集。让我们测量前三个图像帧中的所有对象(大约 300 个细胞核)。
from dask_image import ndmeasure
# Create labelled mask
label_images, num_features = ndmeasure.label(binary_images[:3], structuring_element)
index = np.arange(num_features - 1) + 1 # [1, 2, 3, ...num_features]
这是从我们的掩膜生成的标签图像的截图。
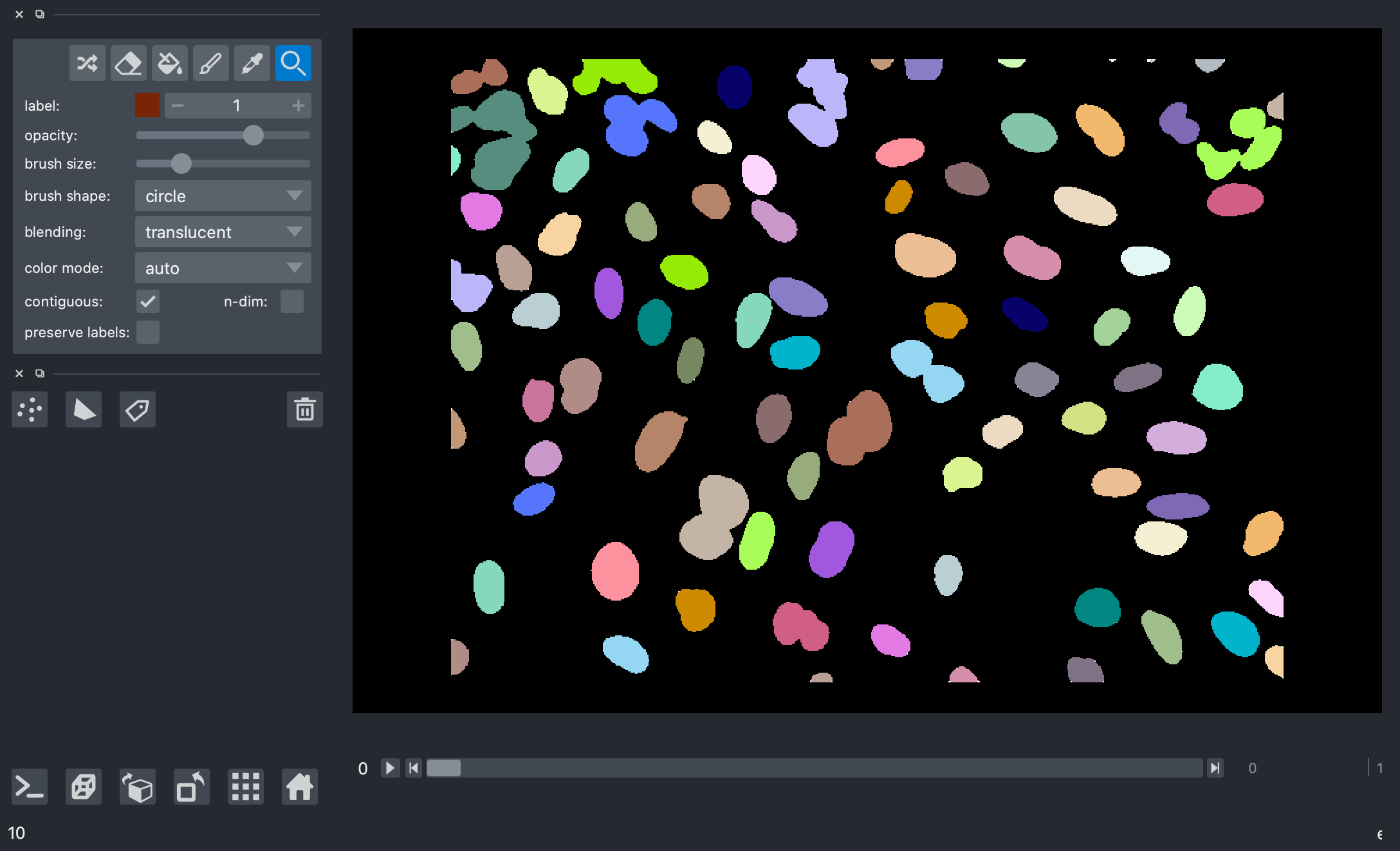
>>> print("Number of nuclei:", num_features.compute())
Number of nuclei: 271
测量图像中的对象
dask-image 的 ndmeasure 子包包含了许多不同的测量函数。在本例中,我们将选择测量
- 每个对象的像素面积,以及
- 每个对象的平均强度。
area = ndmeasure.area(images[:3], label_images, index)
mean_intensity = ndmeasure.mean(images[:3], label_images, index)
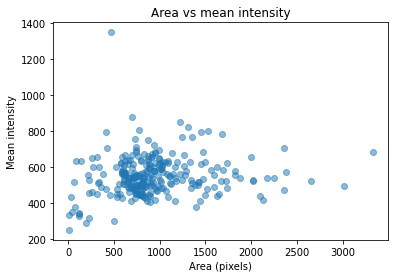
运行计算并绘制结果
import matplotlib.pyplot as plt
plt.scatter(area, mean_intensity, alpha=0.5)
plt.gca().update(dict(title="Area vs mean intensity", xlabel='Area (pixels)', ylabel='Mean intensity'))
plt.show()
自定义函数
如果想做 dask-image API 中不包含的事情怎么办?我们可以使用几种选项来编写自定义函数。
- dask map_overlap / map_blocks
- dask delayed
- scikit-image apply_parallel()
Dask map_overlap 和 map_blocks
Dask 数组的 map_overlap 和 map_blocks 是构建 dask-image 中大多数函数的基础。你也可以自己使用它们。它们将对 Dask 数组中的每个块应用一个函数。
import dask.array as da
def my_custom_function(args):
# ... does something really cool
result = da.map_overlap(my_custom_function, my_dask_array, args)
你可以在这里阅读更多关于 重叠计算 的信息。
Dask delayed
如果想要更灵活和细粒度地控制计算,可以使用 Dask delayed。你可以在 Dask delayed 教程 开始学习。
scikit-image apply_parallel 函数
如果你经常使用 Python 进行图像处理,很可能已经在用 scikit-image。我想提请你注意 scikit-image 中提供的 apply_parallel 函数。它使用了 map-overlap,非常有用。
它不仅在大数据时有用,在数据能放入内存但计算内存密集型时也很有用。这可能会导致超出可用内存,而 apply_parallel 在这些情况下也非常出色。
扩展计算规模
当你想从笔记本电脑扩展到超级计算集群时,可以使用 dask-distributed 来处理。
from dask.distributed import Client
# Setup a local cluster
# By default this sets up 1 worker per core
client = Client()
client.cluster
参阅这里的文档,了解如何为你的系统进行设置。
额外内容:在 GPU 上使用数组
我们最近一直在为 dask-image 添加 GPU 支持。
我们可以使用名为 CuPy 的库添加 GPU 支持。CuPy 是一个具有类似 numpy API 的数组库,由 NVIDIA CUDA 加速。我们可以拥有包含 cupy 块而不是 numpy 块的 Dask 数组。这篇 博文 解释了 GPU 加速的好处,并提供了在 CPU、单个 GPU 和多个 GPU 上进行计算的一些基准测试。
dask-image 中可用的 GPU 支持
从 dask-image 0.6.0 版本开始,六个子包中有四个支持 GPU 数组
- imread
- ndfilters
- ndinterp
- ndmorph
尚未获得 GPU 支持的 dask-image 子包有:
- ndfourier
- ndmeasure
我们希望将来能为这些子包添加 GPU 支持。
示例
这里是使用 Dask 在 CPU 上进行图像卷积的示例
# CPU example
import numpy as np
import dask.array as da
from dask_image.ndfilters import convolve
s = (10, 10)
a = da.from_array(np.arange(int(np.prod(s))).reshape(s), chunks=5)
w = np.ones(a.ndim * (3,), dtype=np.float32)
result = convolve(a, w)
result.compute()
这里是使用 Dask 在 GPU 上进行图像卷积的相同示例。唯一需要改变的是输入数组的类型。
# Same example moved to the GPU
import cupy # <- import cupy instead of numpy (version >=7.7.0)
import dask.array as da
from dask_image.ndfilters import convolve
s = (10, 10)
a = da.from_array(cupy.arange(int(cupy.prod(cupy.array(s)))).reshape(s), chunks=5) # <- cupy dask array
w = cupy.ones(a.ndim * (3,)) # <- cupy array
result = convolve(a, w)
result.compute()
在同一次计算中,不能混合使用 CPU 上的数组和 GPU 上的数组。这就是为什么在上面的第二个示例中,权重 w 必须是 cupy 数组。
此外,可以在 CPU 和 GPU 之间传输数据。因此,在 GPU 加速效果大于数据传输成本的情况下,这样做可能很有用。
将图像读入 GPU
通常,我们想通过读取存储在磁盘上的图像数据来开始图像处理。可以使用 imread 函数并带上 arraytype=cupy 关键字参数来完成此操作。
from dask_image.imread import imread
images = imread('data/BBBC039/images/*.tif')
images_on_gpu = imread('data/BBBC039/images/*.tif', arraytype="cupy")
如何参与
使用 Dask 创建和分享你自己的分割或图像处理工作流程(加入当前关于分割的讨论 或 在此提议新的博文主题)
贡献力量,为 dask-image 添加 GPU 支持:https://github.com/dask/dask-image/issues/133
博客评论由 Disqus 提供支持