骨架分析
作者:Genevieve Buckley
执行摘要
在这篇博文中,我们展示了如何使用 Dask 修改骨架网络分析,使其在内存受限的环境下工作(例如:在你的笔记本电脑上)。这使得它更容易访问:可以在小型笔记本电脑上运行,而无需访问超级计算集群。示例代码也在此提供。
目录
骨架结构无处不在
许多生物结构具有骨架或网络状的形状。我们在各种地方都能看到它们,包括
- 血管分支
- 气道分支
- 大脑中的神经元网络
- 植物的根结构
- 叶脉
- ……以及更多
分析这些骨架的结构可以为我们提供关于该系统生物学的重要信息。
科学问题
对于这篇博文,我们将研究肺内的血管。这些数据由 Marcus Kitchen、Andrew Stainsby 及其协作团队与我们共享。
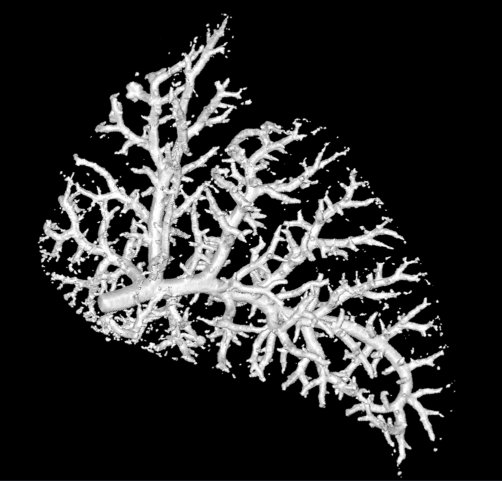
这个研究小组专注于肺部发育。我们想比较健康肺部的血管与疝气模型肺部的血管。在疝气模型中,肺部发育不全,受到挤压,且体积更小。
计算问题
这些图像体大约是 1000x1000x1000 像素。这看起来不大,但考虑到处理分析所需的高内存消耗,在笔记本电脑上运行时会崩溃。
如果你内存不足,有两种可能的方法
-
获取更多内存。在更大的计算机上运行,或将其转移到超级计算集群。这样做的好处是你无需重写代码,但这确实需要访问更强大的计算机硬件。
-
管理你现有的内存。Dask 在这方面表现出色。如果我们使用 Dask 并对数组进行合理的分块,我们可以有效地管理内存,从而不会达到内存上限并崩溃。这样做的好处是你无需购买更多计算机硬件,但这确实需要重写一些代码。
我们的方法
我们采取了第二种方法,使用 Dask,以便在内存受限的小型笔记本电脑上运行分析而不会崩溃。这使得它对更多人来说更容易访问。
所有图像预处理步骤将使用 dask-image 以及 scikit-image 的 skeletonize 函数完成。
我们使用 skan 作为我们分析管道的骨干。skan 是一个用于骨架图像分析的库。给定一个骨架图像,它可以描述分支的统计信息。为了提高速度,该库使用 numba 进行了加速(如果你好奇,可以在这次演讲及其相关笔记本中了解更多)。
此处提供了一个包含骨架分析完整细节的示例笔记本。你可以继续阅读以了解其亮点。
结果
健康肺和疝气肺血管分支的统计数据显示两者之间存在明显差异。
最显著的是血管分支数量的差异。疝气肺的血管分支数量不到健康肺的 40%。
血管尺寸也存在定量差异。这里是一个小提琴图,显示了每个血管分支起点和终点之间距离的分布。我们可以看到,总体而言,疝气肺中的血管分支起点和终点距离更近。这与我们预期的结果一致,因为健康肺比疝气模型中的肺发育更充分,而且疝气将该肺压缩到了更小的整体空间。
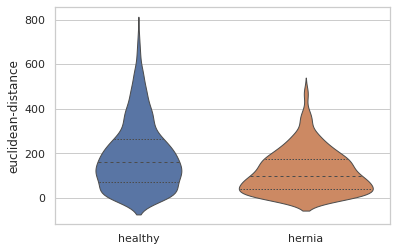
编辑:这篇博文之前将欧几里得距离小提琴图描述为测量血管厚度。这是不正确的,并且在发布前的评审过程中没有发现此错误。此博文已更新,正确描述了 euclidean-distance 测量是分支起点和终点之间的距离,就像在这两点之间拉紧一条线一样。另一种测量 branch-length 描述了包括所有弯曲曲折在内的总分支长度。
局限性
我们依赖一个重要假设:一旦骨架化,减少的非零像素数据将能够放入内存。虽然对于这种大小的数据集(裁剪后的兔子肺数据集大约是 1000 x 1000 x 1000 像素)来说,这是成立的,但对于更大的数据可能不成立。
在我们的原型工作流程中,Dask 计算也在几个点被触发。理想情况下,所有计算都应延迟到最后阶段。
遇到的问题
这个项目最初打算是一个快速简单的项目。真是flag!
我本来想做的是将图像数据放入 Dask 数组,然后使用 map_overlap 函数进行图像滤波、阈值处理、骨架化和骨架分析。但我很快发现,虽然图像滤波、阈值处理和骨架化工作得很好,但骨架分析步骤存在一些问题
-
Dask 的 map_overlap 函数不能很好地处理来自不同图像块的不规则或非均匀形状的结果,并且……
-
skan 库中的内部函数编写方式与分布式计算不兼容。
我们如何解决它们
问题 1:scikit-image 的 skeletonize 函数由于内存不足而崩溃
由 scikit-image 提供的 skeletonize 函数非常占用内存,并在 16GB RAM 的笔记本电脑上崩溃。
我们通过以下方法解决了这个问题
- 使用
dask-image imread将图像数据放入 Dask 数组中, - 对 Dask 数组进行重新分块。我们需要将分块形状从 2D 切片更改为小的立方体体积,以便下一步计算更高效。我们可以选择分块的总体大小,使其能够保持在 skeletonize 所需的内存阈值以下。
- 最后,我们使用
map_overlap函数在 Dask 数组块上运行skeletonize函数。通过限制数组块的大小,我们保持在内存阈值以下!
问题 2:Dask 数组块产生不规则或非均匀输出
骨架分析函数将为每个图像块返回长度不规则或非均匀的结果。这并不奇怪,因为不同的块在我们的骨架形状中会有不同数量的非零像素。
在使用 Dask 数组时,有两个非常常用的函数:map_blocks 和 map_overlap。当我们尝试使用 map_blocks 与 map_overlap 处理具有不规则输出的函数时,会发生以下情况。
import dask.array as da
import numpy as np
x = da.ones((100, 10), chunks=(10, 10))
def foo(a): # our dummy analysis function
random_length = np.random.randint(1, 7)
return np.arange(random_length)
使用 map_blocks 时,一切正常
result = da.map_blocks(foo, x, drop_axis=1)
result.compute() # this works well
但是如果我们函数 foo 需要一些重叠才能正确工作,那么就会遇到问题
result = da.map_overlap(foo, x, depth=1, drop_axis=1)
result.compute() # incorrect results
在这里,在结果被连接之前,foo 的第一个和最后一个元素被截掉了,这不是我们想要的!设置关键字参数 trim=False 有助于避免这个问题,但那样我们就会得到一个错误
result = da.map_overlap(foo, x, depth=1, trim=False, drop_axis=1)
result.compute() # ValueError
不幸的是,对于我们来说,在数组块中保留 1 像素的重叠非常重要,这样我们才能判断骨架分支是结束了还是延续到了下一个块。
map_overlap 结果连接回来的方式有些复杂,因此与其深入研究,更直接的解决方案是使用 Dask delayed。 Chris Roat 提供了一个很好的例子,展示了我们如何在列表推导式中使用 Dask delayed,然后用 Dask 进行连接(原始讨论链接)。
import numpy as np
import pandas as pd
import dask
import dask.array as da
import dask.dataframe as dd
x = da.ones((20, 10), chunks=(10, 10))
@dask.delayed
def foo(a):
size = np.random.randint(1,10) # Make each dataframe a different size
return pd.DataFrame({'x': np.arange(size),
'y': np.arange(10, 10+size)})
meta = dd.utils.make_meta([('x', np.int64), ('y', np.int64)])
blocks = x.to_delayed().ravel() # no overlap
results = [dd.from_delayed(foo(b), meta=meta) for b in blocks]
ddf = dd.concat(results)
ddf.compute()
警告:将 meta 关键字参数传递给 from_delayed 函数非常重要。否则,效率会非常低下!
如果未提供 meta 关键字参数,Dask 将尝试自行确定其值。通常这可能是好事,但在列表推导式中,这意味着这些任务在主计算开始之前就会缓慢地按顺序计算,效率极低。由于我们提前知道分析函数会返回什么样的结果(我们只是不知道每组结果的长度),因此可以使用 utils.make_meta 函数来帮助我们。
问题 3:抓取带有重叠部分的图像块
现在我们使用 Dask delayed 来组合我们的骨架分析结果,我们需要自己处理数组块的重叠部分。
我们将通过修改 Dask 的 dask.array.core.slices_from_chunks 函数来实现这一点,使其能够处理重叠。在 Dask 数组的边界需要进行一些特殊处理,以便我们不会尝试切片超出数组边缘。
代码如下所示(gist)
from itertools import product
from dask.array.slicing import cached_cumsum
def slices_from_chunks_overlap(chunks, array_shape, depth=1):
cumdims = [cached_cumsum(bds, initial_zero=True) for bds in chunks]
slices = []
for starts, shapes in zip(cumdims, chunks):
inner_slices = []
for s, dim, maxshape in zip(starts, shapes, array_shape):
slice_start = s
slice_stop = s + dim
if slice_start > 0:
slice_start -= depth
if slice_stop >= maxshape:
slice_stop += depth
inner_slices.append(slice(slice_start, slice_stop))
slices.append(inner_slices)
return list(product(*slices))
现在我们可以切取一个图像块并增加一个额外的重叠像素,我们只需要一种方法对数组中的所有块执行此操作。借鉴此处的块迭代,我们创建了一个类似的迭代器。
block_iter = zip(
np.ndindex(*image.numblocks),
map(functools.partial(operator.getitem, image),
slices_from_chunks_overlap(image.chunks, image.shape, depth=1))
)
meta = dd.utils.make_meta([('row', np.int64), ('col', np.int64), ('data', np.float64)])
intermediate_results = [dd.from_delayed(skeleton_graph_func(block), meta=meta) for _, block in block_iter]
results = dd.concat(intermediate_results)
results = results.drop_duplicates() # we need to drop duplicates because it counts pixels in the overlapping region twice
有了这些结果,我们就能创建稀疏骨架图了。
问题 4:使用 skan 计算摘要统计
骨架分支统计可以使用 skan summarize 函数计算。这里的问题是该函数需要一个 Skeleton 对象实例,但是初始化 Skeleton 对象会调用与分布式分析不兼容的方法。
我们将通过以下方法解决这个问题:首先使用一个微小的虚拟数据集初始化一个 Skeleton 对象实例,然后用我们的真实结果覆盖骨架对象的属性。这是一个权宜之计,但它能让我们达到目标:为我们的大数据集计算摘要分支统计。
首先,我们使用虚拟数据创建一个 Skeleton 对象实例
from skan._testdata import skeleton0
skeleton_object = Skeleton(skeleton0) # initialize with dummy data
然后我们用之前计算的结果覆盖属性
skeleton_object.skeleton_image = ...
skeleton_object.graph = ...
skeleton_object.coordinates
skeleton_object.degrees = ...
skeleton_object.distances = ...
...
最后,我们可以计算摘要分支统计
from skan import summarize
statistics = summarize(skel_obj)
statistics.head()
| 骨架 ID | 源节点 ID | 目标节点 ID | 分支距离 | 分支类型 | 平均像素值 | 像素值标准差 | 图像坐标源 0 | 图像坐标源 1 | 图像坐标源 2 | 图像坐标目标 0 | 图像坐标目标 1 | 图像坐标目标 2 | 坐标源 0 | 坐标源 1 | 坐标源 2 | 坐标目标 0 | 坐标目标 1 | 坐标目标 2 | 欧几里得距离 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 1 | 1 | 2 | 1 | 2 | 0.474584 | 0.00262514 | 22 | 400 | 595 | 22 | 400 | 596 | 22 | 400 | 595 | 22 | 400 | 596 | 1 |
| 1 | 2 | 3 | 9 | 8.19615 | 2 | 0.464662 | 0.00299629 | 37 | 400 | 622 | 43 | 392 | 590 | 37 | 400 | 622 | 43 | 392 | 590 | 33.5261 |
| 2 | 3 | 10 | 11 | 1 | 2 | 0.483393 | 0.00771038 | 49 | 391 | 589 | 50 | 391 | 589 | 49 | 391 | 589 | 50 | 391 | 589 | 1 |
| 3 | 5 | 13 | 19 | 6.82843 | 2 | 0.464325 | 0.0139064 | 52 | 389 | 588 | 55 | 385 | 588 | 52 | 389 | 588 | 55 | 385 | 588 | 5 |
| 4 | 7 | 21 | 23 | 2 | 2 | 0.45862 | 0.0104024 | 57 | 382 | 587 | 58 | 380 | 586 | 57 | 382 | 587 | 58 | 380 | 586 | 2.44949 |
statistics.describe()
| 骨架 ID | 源节点 ID | 目标节点 ID | 分支距离 | 分支类型 | 平均像素值 | 像素值标准差 | 图像坐标源 0 | 图像坐标源 1 | 图像坐标源 2 | 图像坐标目标 0 | 图像坐标目标 1 | 图像坐标目标 2 | 坐标源 0 | 坐标源 1 | 坐标源 2 | 坐标目标 0 | 坐标目标 1 | 坐标目标 2 | 欧几里得距离 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 计数 | 1095 | 1095 | 1095 | 1095 | 1095 | 1095 | 1095 | 1095 | 1095 | 1095 | 1095 | 1095 | 1095 | 1095 | 1095 | 1095 | 1095 | 1095 | 1095 | 1095 |
| 平均值 | 2089.38 | 11520.1 | 11608.6 | 22.9079 | 2.00091 | 0.663422 | 0.0418607 | 591.939 | 430.303 | 377.409 | 594.325 | 436.596 | 373.419 | 591.939 | 430.303 | 377.409 | 594.325 | 436.596 | 373.419 | 190.13 |
| 标准差 | 636.377 | 6057.61 | 6061.18 | 24.2646 | 0.0302199 | 0.242828 | 0.0559064 | 174.04 | 194.499 | 97.0219 | 173.353 | 188.708 | 96.8276 | 174.04 | 194.499 | 97.0219 | 173.353 | 188.708 | 96.8276 | 151.171 |
| 最小值 | 1 | 1 | 2 | 1 | 2 | 0.414659 | 6.79493e-06 | 22 | 39 | 116 | 22 | 39 | 114 | 22 | 39 | 116 | 22 | 39 | 114 | 0 |
| 25% | 1586 | 6215.5 | 6429.5 | 1.73205 | 2 | 0.482 | 0.00710439 | 468.5 | 278.5 | 313 | 475 | 299.5 | 307 | 468.5 | 278.5 | 313 | 475 | 299.5 | 307 | 72.6946 |
| 50% | 2431 | 11977 | 12010 | 16.6814 | 2 | 0.552626 | 0.0189069 | 626 | 405 | 388 | 627 | 410 | 381 | 626 | 405 | 388 | 627 | 410 | 381 | 161.059 |
| 75% | 2542.5 | 16526.5 | 16583 | 35.0433 | 2 | 0.768359 | 0.0528814 | 732 | 579 | 434 | 734 | 590 | 432 | 732 | 579 | 434 | 734 | 590 | 432 | 265.948 |
| 最大值 | 8034 | 26820 | 26822 | 197.147 | 3 | 1.29687 | 0.357193 | 976 | 833 | 622 | 976 | 841 | 606 | 976 | 833 | 622 | 976 | 841 | 606 | 737.835 |
成功!
我们已经使用 Dask 实现了分布式骨架分析。您可以在此处查看包含骨架分析完整细节的示例笔记本。
下一步是什么?
一个好的下一步是修改 skan 库代码,使其直接支持分布式骨架分析。
你能如何提供帮助
如果你想参与进来,有几个选择
- 尝试在你自己的数据上进行类似的分析。包含完整示例代码的笔记本在此提供。你可以在 Dask slack 或 twitter 上分享或提问。
- 帮助为 skan 添加分布式骨架分析支持。请前往 skan issues 页面,如果你想加入,请留言。
博客评论由 Disqus 提供支持